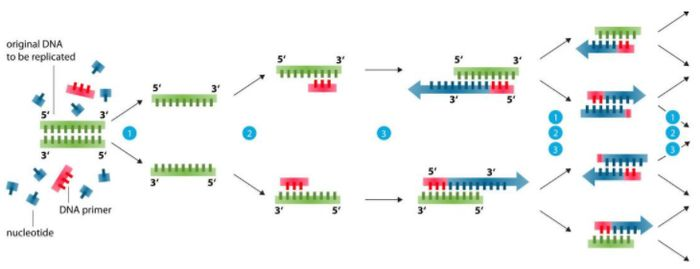
Primer designbas (99% problem kan lösas)
1. Primerlängd: Läroboken kräver 15-30bp, vanligtvis cirka 20bp.Det faktiska tillståndet är bättre att vara 18-24bp för att säkerställa specificiteten, men ju längre desto bättre, för lång primer kommer också att minska specificiteten och minska utbytet.
2. Primeramplifieringsintervall: 200-500 bp är lämpligt, och fragmentet kan expanderas till 10 kb under specifika förhållanden.
3. Primerbas: Innehållet av G+C bör vara 40-60%, för lite G+C-förstärkningseffekt är inte bra, för mycket G+C är lätt att visa ospecifika band.ATGC fördelas bäst slumpmässigt och undviker kluster med mer än 5 purin- eller pyrimidinnukleotider.Multi-gc för 5'-änden och mellansekvenser för att öka stabiliteten, undvika rik GC vid 3'-änden, ingen GC för de sista 3 baserna eller ingen GC för 3 av de sista 5 baserna.
4. Undvik sekundär struktur i primrar och undvik komplementering mellan två primrar, speciellt komplementering i 3'-änden, annars kommer primerdimer att bildas och icke-specifika amplifierade band genereras.
5. Baserna vid 3'-änden av primrarna, särskilt de sista och näst sista baserna, bör paras strikt för att undvika PCR-fel på grund av oparade terminala baser.
6. Primers har eller kan tillsättas med lämpliga klyvningsställen, och den amplifierade målsekvensen bör företrädesvis ha lämpliga klyvningsställen, vilket är mycket fördelaktigt för klyvningsanalys eller molekylär kloning.
7. Specificitet för primrar: primrar bör inte ha någon uppenbar homologi med andra sekvenser i nukleinsyrasekvensdatabasen.
8. Lär dig att använda programvara: PP5, Oligo6, DNAstar, Vector NTI, primer3 (Denna onlinedesign fungerar bäst).
Ovanstående innehåll kan lösa åtminstone 99 % av primerdesignproblemen.
Kontrollera detaljerna i primerdesign
1. Primerlängd
Generell primerlängd är 18~30 baser.I allmänhet är den viktigaste faktorn som bestämmer primerns hybridiseringstemperatur längden på primern.Glödgningstemperaturen för primer väljs i allmänhet (Tm-värde -5℃), och vissa använder direkt Tm-värdet.Följande formler kan användas för att grovt beräkna glödgningstemperaturen för primers.
När primerns längd är mindre än 20bp: [4(G+C)+2(A+T)]-5℃
När primerns längd är större än 20 bp: 62,3℃+0,41℃(%GC)-500/längd-5℃
Dessutom kan många program också användas för att beräkna glödgningstemperaturen, beräkningsprincipen kommer att vara annorlunda, så ibland kan det beräknade värdet ha ett litet gap.För att optimera PCR-reaktioner används de kortaste primrarna som säkerställer hybridiseringstemperaturer på inte mindre än 54 ℃ för bästa effektivitet och specificitet.
Sammantaget ökar primerspecificiteten med en faktor fyra för varje ytterligare nukleotid, så att den minsta primerlängden för de flesta tillämpningar är 18 nukleotider.Den övre gränsen för primerlängden är inte särskilt viktig, främst relaterad till reaktionseffektiviteten.På grund av entropi, ju längre primern är, desto lägre hastighet med vilken den hybridiserar för att binda till mål-DNA för att bilda en stabil dubbelsträngad mall för DNA-polymeras att binda.
När man använder programvara för att designa primers, kan längden på primers bestämmas av TM-värdet i sin tur, speciellt för primers av fluorescens kvantitativ PCR, TM=60℃ eller så bör kontrolleras.
2.GC-innehåll
I allmänhet är innehållet av G+C i primersekvenser 40%~60%, och GC-innehåll och Tm-värde för ett par primrar bör koordineras.Om primern har en allvarlig GC- eller AT-tendens kan lämplig mängd A, T eller G och C-svans läggas till 5'-änden av primern.
3. Glödgningstemperatur
Glödgningstemperaturen bör vara 5 ℃ lägre än avkedjetemperaturen.Om antalet primerbaser är litet kan hybridiseringstemperaturen höjas på lämpligt sätt, vilket kan öka specificiteten för PCR.Om antalet baser är stort kan glödgningstemperaturen reduceras på lämpligt sätt.Glödgningstemperaturskillnaden mellan ett par primrar på 4 ℃ ~ 6 ℃ kommer inte att påverka PCR-utbytet, men idealiskt är glödgningstemperaturen för ett par primrar densamma, vilket kan variera mellan 55 ℃ ~ 75 ℃.
4. Undvik det sekundära strukturområdet för amplifieringsmallen
Det är bäst att undvika mallens sekundära strukturregion när man väljer det amplifierade fragmentet.Den stabila sekundära strukturen för målfragmentet kan förutsägas och uppskattas av relevant datormjukvara, vilket är användbart för mallval.Experimentella resultat visar att expansionen ofta misslyckas när den fria energin (△G) i regionen som ska expanderas är mindre än 58,6 lkJ/mol.
5. Felmatchning med mål-DNA
När den amplifierade mål-DNA-sekvensen är stor, kan en primer binda till flera delar av mål-DNA:t, vilket resulterar i att flera band uppträder i resultatet.Den här gången är det nödvändigt att använda BLAST mjukvarutestning, webbplats:http://www.ncbi.nlm.nih.gov/BLAST/.Välj Justera två sekvenser (bl2seq).
Att klistra in primersekvenser till zon 1 och mål-DNA-sekvenser till zon 2 är utbytbart och BLAST beräknar komplementära, antisense och andra möjligheter, så att användarna inte behöver märka om båda kedjorna är avkänningskedjor.Du kan också ange GI-numret om du känner till GI-numret för sekvensen i databasen, så du behöver inte klistra in en stor del av sekvensen.Klicka slutligen på Justera vid 3 för att se om primern har flera homologa ställen i mål-DNA:t.
6. Primerterminal
3'-änden av primern är där förlängningen börjar, så det är viktigt att förhindra att missmatchningar börjar där.3'-änden bör inte vara mer än 3 på varandra följande G eller C, eftersom detta kommer att orsaka att primern av misstag triggas i G+C-anrikningssekvensregionen.3'-änden kan inte bilda någon sekundär struktur, förutom i speciella PCR-reaktioner (AS-PCR) kan 3'-änden av primern inte vara felmatchad.Till exempel, om den kodande regionen amplifieras, bör 3'-änden av primern inte avslutas vid kodonets tredje position, eftersom kodonets tredje position är benägen att degenerera, vilket kommer att påverka specificiteten och effektiviteten av amplifieringen.När du använder annexationsprimrar, se tabellen för kodonanvändning, var uppmärksam på biologiska preferenser, använd inte annexationsprimrar i 3′-änden och använd en högre koncentration av primers (1uM-3uM).
7. Sekundär struktur av primers
Själva primrarna bör inte ha komplementära sekvenser, annars kommer själva primrarna att vikas till hårnålsstrukturer, och denna sekundära struktur kommer att påverka bindningen av primers och mallar på grund av steriskt hinder.Om artificiell bedömning används bör de kontinuerliga komplementära baserna för själva primrarna inte vara större än 3bp.Det bör inte finnas någon komplementaritet mellan de två primrarna, särskilt den komplementära överlappningen av 3'-änden bör undvikas för att förhindra bildandet av primerdimerer.I allmänhet bör det inte finnas mer än 4 på varandra följande baser homologi eller komplementaritet mellan ett par primrar.
8. Lägg till markörer eller loki
5'-änden har liten effekt på amplifieringsspecificiteten och kan därför modifieras utan att påverka amplifieringsspecificiteten.Modifieringen av primer 5'-änden innefattade: tillsats av enzymrestriktionsställe;Märkt biotin, fluorescens, digoxin, Eu3+, etc. Introducera proteinbindande DNA-sekvenser;Införa mutationsställen, infoga och sakna mutationssekvenser och introducera promotorsekvenser etc. De extra baserna kommer mer eller mindre att påverka effektiviteten av amplifiering och öka chansen för primerdimerbildning, men vissa eftergifter måste göras för nästa steg.Ytterligare sekvenser som inte existerar på målsekvensen, såsom restriktionsställen och promotorsekvenser, kan läggas till 5'-änden av primern utan att specificiteten påverkas.Dessa sekvenser ingår inte i beräkningen av primer Tm-värden, men bör testas för komplementaritet och intern sekundär struktur.
9. Subkloner
För det mesta är PCR bara preliminär kloning, och då måste vi subklona målfragmentet till olika vektorer, så vi måste designa ytterligare baser för nästa operation i PCR-steget.
Några sekvenser utformade för subkloning sammanfattas nedan.
Restriktionsendonukleasrestriktionsställe tillsattes
Att lägga till enzymrestriktionsställen är den mest använda metoden för subkloning av PCR-produkter.I allmänhet är klyvningsstället sex baser, utöver 5'-änden av klyvningsstället måste man lägga till 2 ~ 3 skyddande baser.Men antalet skyddande baser som krävs av olika enzymer är olika.Till exempel kräver SalⅠ ingen skyddande bas, EcoRⅤ kräver 1 skyddande bas, NotⅠ kräver 2 skyddande baser och Hind Ⅲ kräver 3 skyddande baser.
LIC lägger till svansen
Det fullständiga namnet på LIC är Ligation-Independent cloning, en kloningsmetod som uppfunnits av Navogen specifikt för sin del av pET-vektorn.pET-bäraren framställd med LIC-metoden har icke-komplementära 12-15 bas enkelsträngade klibbiga ändar, som kompletterar motsvarande klibbiga ändar på målinsättningsfragmentet.För amplifieringsändamål bör primerns 5'-sekvens för det insatta fragmentet komplettera LIC-vektorn.3′→5′ extranect-aktiviteten av T4 DNA-polymeras kan bilda en enkelsträngad klibbig ände på det infogade fragmentet efter en kort tid.Eftersom produkten endast kan bildas från den ömsesidiga hybridiseringen av det preparerade insättningsfragmentet och vektorn, är denna metod mycket snabb och effektiv, och den är riktad kloning.
Riktad TA klon lägga till svans
TA-kloning kunde inte rikta fragmentet in i en vektor, så senare introducerade Invitrogen en vektor som kunde rikta in sig på kloning, som innehöll fyra framträdande bas GTGGS i ena änden.Därför, vid utformningen av PCR-primrar, bör komplementära sekvenser läggas till i enlighet därmed, så att fragment kan "orienteras".
Om du har ont om tid kan du prova direkt syntes, kombinera genen med vektorn, vilket är vad vi kallar ET-gensyntes hos musekulare.
D. In-Fusion kloningsmetod
Inget ligas krävs, ingen lång reaktion krävs.Så länge som en sekvens i båda ändarna av den linjäriserade vektorn införs. Vid design av primrar, sedan tillsätts PCR-produkten och den linjäriserade vektorn till infusionsenzymlösningen innehållande BSA och placeras i rumstemperatur i en halvtimme, kan transformationen utföras.Denna metod är särskilt lämplig för konvertering av stora volymer.
10. Sammanfoga primer
Ibland är endast begränsad sekvensinformation känd om primerdesign.Till exempel, om endast aminosyrasekvensen är känd, kan den sammanslagna primern utformas.En sammanslagningsprimer är en blandning av olika sekvenser som representerar alla olika basmöjligheter som kodar för en enda aminosyra.För att öka specificiteten kan du hänvisa till kodonanvändningstabellen för att minska annektering enligt basanvändningspreferenser för olika organismer.Hypoxantin kan paras ihop med alla baserna för att reducera glödgningstemperaturen för primern.Använd inte de bifogade baserna vid 3′-änden av primern eftersom hybridiseringen av de sista 3 baserna vid 3′-änden är tillräcklig för att initiera PCR på fel ställe.Högre primerkoncentrationer (1μM till 3μM) används eftersom primers i många annexationsblandningar inte är specifika för målmallen.
PCR-råvarorkontrollera
1. Grundfärgsmängd
Koncentrationen av varje primer är 0,1 ~ 1 umol eller 10 ~ 100 pmol.Det är bättre att producera det önskade resultatet med den lägsta mängden primer.Hög koncentration av primer kommer att orsaka felparning och ospecifik amplifiering och ökar chansen att bilda dimerer mellan primrar.
2. Primerkoncentration
Koncentrationen av primers påverkar specificiteten.Den optimala primerkoncentrationen är vanligtvis mellan 0,1 och 0,5 μM.Högre primerkoncentrationer leder till amplifiering av ospecifika produkter.
3. Glödgningstemperatur för primer
En annan viktig parameter för primers är smälttemperaturen (Tm).Detta är temperaturen när 50 % av primrarna och de komplementära sekvenserna representeras som dubbelsträngade DNA-molekyler.Tm krävs för att ställa in PCR-glödgningstemperaturen.Idealiskt är hybridiseringstemperaturen tillräckligt låg för att säkerställa effektiv hybridisering av primrarna med målsekvensen, men tillräckligt hög för att minska ospecifik bindning.Rimlig glödgningstemperatur från 55 ℃ till 70 ℃.Glödgningstemperaturen är i allmänhet inställd på 5 ℃ lägre än Tm för primern.
Det finns flera formler för inställning av Tm, som varierar mycket beroende på vilken formel som används och sekvensen av primrar.Eftersom de flesta formler ger ett uppskattat Tm-värde är alla glödgningstemperaturer bara en utgångspunkt.Specificiteten kan förbättras genom att analysera flera reaktioner som progressivt höjer glödgningstemperaturen.Börja under den uppskattade Tm-5 ℃ och öka gradvis glödgningstemperaturen i ett steg på 2 ℃.Högre glödgningstemperatur kommer att minska bildningen av primerdimerer och icke-specifika produkter.För bästa resultat bör de två primrarna ha ungefärliga Tm-värden.Om Tm-skillnaden för primerpar är mer än 5 ℃, kommer primrarna att visa en signifikant felaktig start genom att använda en lägre glödgningstemperatur i cykeln.Om de två primrarna Tm är olika, ställ in glödgningstemperaturen till 5 ℃ lägre än den lägsta Tm.Alternativt, för att öka specificiteten, kan fem cykler utföras först vid glödgningstemperaturer utformade för högre Tm, följt av de återstående cyklerna vid glödgningstemperaturer utformade för lägre Tm.Detta gör att en delkopia av destinationsmallen kan erhållas under snäva förhållanden.
4. Primerrenhet och stabilitet
Standardrenheten för anpassade primers är tillräcklig för de flesta PCR-tillämpningar.Avlägsnandet av bensoyl- och isobutylylgrupper genom avsaltning är minimalt och stör därför inte PCR.Vissa tillämpningar kräver rening för att avlägsna eventuella icke-fullängdssekvenser i syntesprocessen.Dessa trunkerade sekvenser uppstår eftersom effektiviteten av DNA-synteskemin inte är 100 %.Detta är en cirkulär process som använder upprepade kemiska reaktioner då varje bas läggs till för att göra DNA från 3′ till 5′.Du kan misslyckas i båda cyklerna.Längre primrar, särskilt de som är större än 50 baser, har en stor andel trunkerade sekvenser och kan kräva rening.
Utbytet av primers påverkas av effektiviteten hos syntetisk kemi och reningsmetod.Biofarmaceutiska företag, såsom Cytology och Shengong, använder alla en minsta OD-enhet för att säkerställa den totala produktionen av oligonukleosid.Anpassade primers levereras i torr pulverform.Det är bäst att återupplösa primrarna i TE så att den slutliga koncentrationen är 100μM.TE är bättre än avjoniserat vatten eftersom vattnets pH ofta är surt och kommer att orsaka hydrolys av oligonukleosider.
Stabiliteten hos primers beror på lagringsförhållandena.Torrt pulver och lösta primers bör förvaras vid -20 ℃.Primers upplösta i TE vid koncentrationer över 10 μM kan lagras stabilt vid -20 ℃ i 6 månader, men kan endast förvaras i rumstemperatur (15 ℃ till 30 ℃) i mindre än 1 vecka.Torrpulverprimers kan förvaras vid -20 C i minst 1 år och i rumstemperatur (15 C till 30 C) i upp till 2 månader.
5. Enzymer och deras koncentrationer
För närvarande är det Taq DNA-polymeras som används i grunden det gentekniska enzymet som syntetiseras av koliforma bakterier.Mängden enzym som behövs för att katalysera en typisk PCR-reaktion är cirka 2,5 U (avser den totala reaktionsvolymen på 100 ul).Om koncentrationen är för hög kan det leda till ospecifik amplifiering;om koncentrationen är för låg kommer mängden syntetisk produkt att minska.
6. Kvalitet och koncentration av dNTP
Kvaliteten på dNTP är nära relaterad till koncentrationen och effektiviteten av PCR-amplifiering.dNTP-pulvret är granulärt och dess variation förlorar sin biologiska aktivitet om det förvaras felaktigt.dNTP-lösningen är sur och bör användas i hög koncentration, med 1M NaOH eller 1M Tris.HCL-buffertlösning för att justera dess PH till 7,0 ~ 7,5, liten mängd underförpackningar, fryst lagring vid -20 ℃.Flera frys-upptining kommer att försämra dNTP.Vid PCR-reaktion bör dNTP vara 50 ~ 200umol/L.Särskilt bör uppmärksamhet ägnas åt att koncentrationen av de fyra DNTPS bör vara lika (lika mol beredning).Om koncentrationen av någon av dem skiljer sig från de andra (högre eller lägre), kommer missmatchning att orsakas.För låg koncentration kommer att minska utbytet av PCR-produkter.dNTP kan kombineras med Mg2+ och minska koncentrationen av fritt Mg2+.
7. Mall (målgen) nukleinsyra
Mängden och reningsgraden av mallnukleinsyra är en av nyckellänkarna för framgång eller misslyckande med PCR.De traditionella DNA-reningsmetoderna använder vanligtvis SDS och proteas K för att smälta och kassera prover.Huvudfunktionerna för SDS är: lösa upp lipider och proteiner på cellmembranet, och på så sätt förstöra cellmembranet genom att lösa upp membranproteiner, och dissociera kärnproteiner i cellen, SDS kan också kombineras med proteiner och fällas ut;Proteas K kan hydrolysera och smälta proteiner, särskilt histoner bundna med DNA, och sedan använda organiskt lösningsmedel fenol och kloroform för att extrahera proteiner och andra cellkomponenter, och använda etanol eller isopropylalkohol för att fälla ut nukleinsyra.Den extraherade nukleinsyran kan användas som en mall för PCR-reaktioner.För allmänna kliniska detektionsprover kan en snabb och enkel metod användas för att lösa upp celler, lysera patogener, smälta och ta bort proteiner från kromosomer till fria målgener, och direkt användas för PCR-amplifiering.RNA-mallextraktion använder vanligtvis guanidinisotiocyanat- eller proteas K-metoden för att förhindra RNas från att bryta ned RNA.
8.Mg2+ koncentration
Mg2+ har en signifikant effekt på specificiteten och utbytet av PCR-amplifiering.I en allmän PCR-reaktion, när koncentrationen av olika dNTP är 200umol/L, är den lämpliga koncentrationen av Mg2+ 1,5 ~ 2,0 mmol/L.Mg2+-koncentrationen är för hög, reaktionsspecificiteten minskar, ospecifik amplifiering inträffar, för låg koncentration kommer att minska aktiviteten av Taq DNA-polymeras, vilket resulterar i minskningen av reaktionsprodukter.
Magnesiumjoner påverkar flera aspekter av PCR, såsom DNA-polymerasaktivitet, som påverkar utbytet;Ett annat exempel är primer-annealing, som påverkar specificiteten.dNTP och mall binder till magnesiumjon, vilket minskar mängden fri magnesiumjon som krävs för enzymaktivitet.Den optimala magnesiumjonkoncentrationen varierar för olika primerpar och mallar, men en typisk PCR-startkoncentration med 200 μM dNTP är 1,5 mM (notera: För kvantitativ PCR i realtid, använd 3 till 5 mM magnesiumjonlösning med en fluorescerande sond).Högre koncentrationer av fria magnesiumjoner ökar utbytet, men ökar också ospecifik amplifiering och minskar troheten.För att bestämma den optimala koncentrationen utfördes titrering av magnesiumjoner i steg om 0,5 mM från 1 mM till 3 mM.För att minska beroendet av magnesiumjonoptimering kan Platinum Taq DNA-polymeras användas.Platina Taq DNA-polymeras kan bibehålla funktionen över ett bredare intervall av magnesiumjonkoncentrationer än Taq DNA-polymeras och kräver därför mindre optimering.
9. Pcr-främjande tillsatser
Optimering av hybridiseringstemperatur, primerdesign och magnesiumjonkoncentration är tillräcklig för mycket specifik amplifiering av de flesta mallar;Vissa mallar, inklusive de med högt GC-innehåll, kräver dock ytterligare åtgärder.Tillsatser som påverkar smälttemperaturen för DNA ger ett annat sätt att förbättra produktens specificitet och utbyte.Full denaturering av mallen krävs för bästa resultat.
Dessutom förhindrar den sekundära strukturen primerbindning och enzymförlängning.
PCR-tillsatser, inklusive formamid, DMSO, glycerin, betain och PCRx Enhancer Solution, förbättrar amplifieringen.Deras möjliga mekanism är att sänka smälttemperaturen, vilket underlättar hybridiseringen av primrar och hjälper DNA-polymerasförlängningen genom den sekundära strukturregionen.PCRx Solution har andra fördelar.Minimal magnesiumjonoptimering krävs vid användning med Platinum Taq DNA-polymeras och Platinum Pfx DNA-polymeras.Således kombineras platinatekniken med tillsatsen för att öka specificiteten samtidigt som beroendet av den tredje metoden, magnesiumjonoptimering, minskas.För bästa resultat bör koncentrationen av tillsatser optimeras, särskilt DMSO, formamid och glycerol, som hämmar Taq DNA-polymeras.
10. Varmstart
Hotstart PCR är en av de viktigaste metoderna för att förbättra PCR-specificiteten förutom bra primerdesign.Även om den optimala förlängningstemperaturen för Taq DNA-polymeras är 72 ℃, förblir polymeraset aktivt vid rumstemperatur.Således produceras icke-specifika produkter när hålltemperaturen är lägre än glödgningstemperaturen under framställningen av PCR-reaktionen och i början av den termiska cykeln.När de väl bildats förstärks dessa ospecifika produkter effektivt.Hot-start PCR är särskilt effektiv när de platser som används för primerdesign begränsas av platsen för genetiska element, såsom platsriktade mutationer, uttryckskloning eller konstruktion och manipulation av genetiska element som används för DNA-teknik.
En vanlig metod för att begränsa aktiviteten av Taq DNA-polymeras är att bereda PCR-reaktionslösning på is och placera den i en förvärmd PCR-apparat.Denna metod är enkel och billig, men den fullbordar inte enzymets aktivitet och eliminerar därför inte helt amplifieringen av icke-specifika produkter.
Termisk priming fördröjer DNA-syntes genom att hämma en väsentlig komponent tills PCR-apparaten når denatureringstemperatur.De flesta manuella termiska initieringsmetoder, inklusive fördröjd tillsats av Taq DNA-polymeras, är besvärliga, särskilt för tillämpningar med hög genomströmning.Andra termiska grundningsmetoder använder en vaxsköld för att innesluta en viktig komponent, inklusive magnesiumjoner eller enzymer, eller för att fysiskt isolera reaktiva komponenter, såsom mallar och buffertar.Under den termiska cykeln frigörs de olika komponenterna och blandas ihop när vaxet smälter.Liksom den manuella varmstartmetoden är vaxskyddsmetoden besvärlig och utsatt för kontaminering och är inte lämplig för tillämpningar med hög genomströmning.
Platina DNA-polymeras är bekvämt och effektivt för automatisk varmstart-PCR.Platina Taq DNA-polymeras består av rekombinant Taq DNA-polymeras kombinerat med monoklonal antikropp mot Taq DNA-polymeras.Antikropparna formuleras med PCR för att hämma enzymaktivitet under långvarig temperaturhållning.Taq DNA-polymeras frisattes i reaktionen under 94℃-isoleringen av denatureringssteget, vilket återställde full polymerasaktivitet.I motsats till kemiskt modifierat Taq DNA-polymeras för termisk initiering, kräver platinaenzym inte långvarig isolering vid 94 ℃ (10 till 15 minuter) för att aktivera polymeraset.Med PlatinumTaq DNA-polymeras återställdes 90 % av Taq DNA-polymerasaktiviteten efter 2 minuter vid 94 ℃.
11. Nest-PCR
Successiva omgångar av amplifiering med användning av kapslade primrar kan förbättra specificitet och känslighet.Den första omgången är en standardförstärkning på 15 till 20 cykler.En liten del av den initiala amplifieringsprodukten späddes 100 till 1000 gånger och sattes till den andra omgången av amplifiering under 15 till 20 cykler.Alternativt kan den initiala amplifierade produkten storleksbestämmas genom gelrening.En kapslad primer används i den andra omgången av amplifiering, som kan binda till målsekvensen inuti den första primern.Användningen av kapslad PCR minskar möjligheten för amplifiering av flera målställen eftersom det finns få målsekvenser som är komplementära till båda uppsättningarna av primrar.Samma totala antal cykler (30 till 40) med samma primrar amplifierade de ospecifika ställena.Kapslad PCR ökar känsligheten för begränsade målsekvenser (t.ex. sällsynta mrnas) och förbättrar specificiteten för svår PCRS (t.ex. 5' RACE).
12. Fallande PCR
Fallande PCR förbättrar specificiteten genom att använda snäva glödgningsförhållanden för de första cyklerna av PCR.Cykeln startar vid en glödgningstemperatur som är ungefär 5 ℃ högre än den uppskattade Tm, sedan reduceras varje cykel med 1 ℃ till 2 ℃ tills glödgningstemperaturen är under Tm 5 ℃.Endast destinationsmallen med den högsta homologin kommer att amplifieras.Dessa produkter fortsätter att expandera i efterföljande cykler och tränger ut de förstärkta ospecifika produkterna.Descending PCR är användbar för metoder där graden av homologi mellan primer och målmall inte är känd, såsom AFLP DNA-fingeravtryck.
Relaterade PCR-kit
2× PCR HeroTMMix-systemet har högre tolerans mot PCR-hämmare än det vanliga PCR Mix-systemet och klarar enkelt PCR-förstärkning av olika komplexa mallar.Det unika reaktionssystemet och högeffektiva Taq Hero gör att PCR-reaktionen har högre amplifieringseffektivitet, specificitet och känslighet.
Högre förstärkningseffektivitet
Den har 5'→3' DNA-polymerasaktivitet och 5'→3' exonukleasaktivitet, utan 3'→5' exonukleasaktivitet.
Real Time PCR Easyᵀᴹ-SYBR Green I Kit
Specifik-optimerad buffert och hot-start Taq-enzym kan förhindra ospecifik amplifiering och primerdimerbildning
Hög känslighet – kan upptäcka låga kopior av mall
Satsen använder ett unikt Foregene omvänd transkriptionsreagens och Foregene HotStar Taq DNA-polymeras kombinerat med ett unikt reaktionssystem för att effektivt förbättra amplifieringseffektiviteten och specificiteten hos reaktionen.
Posttid: maj-09-2023