Det är välkänt att i den centrala dogmen är RNA den transkriptionella mediatorn mellan DNA och proteinuttryck.Jämfört med detektion av DNA kan detektionen av RNA mer objektivt återspegla genuttrycket i organismer.Experiment som involverar RNA inkluderar: qRT-PCR, RNA-Seq och fusionsgendetektion etc. Baserat på egenskaperna hos RNA i sig (sockerringen av RNA har en fri hydroxylgrupp mer än sockerringen i DNA), kopplat till ett stort antal RNaser i miljön, är RNA mer instabilt och lättare att brytas ned än DNA.Skräp in, skräp ut, om kvaliteten på RNA inte är bra, måste de experimentella resultaten vara otillfredsställande, specifikt manifesteras som felaktiga data eller dålig repeterbarhet.Därför bör mer uppmärksamhet ägnas åt bearbetning av RNA, och länken till kvalitetskontroll är också viktigare för att säkerställa precisionen och noggrannheten hos efterföljande experimentella data.
För kvalitetskontroll av RNA finns det i allmänhet följande vanliga metoder:
- Spektrofotometri
- agarosgelelektrofores
- Agilent Bioanalyzer
- fluorescerande kvantitativ PCR i realtid
- Qubit fluorescerande färgningsmetod
01 Spektrofotometri
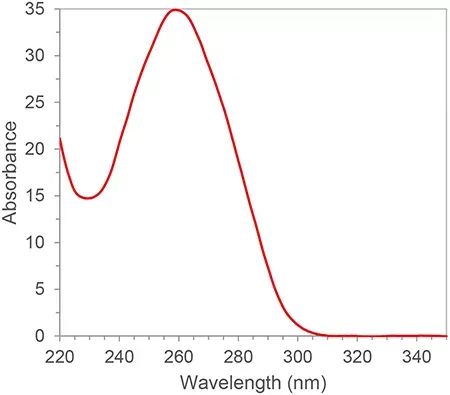
RNA har konjugerade dubbelbindningar och har en absorptionstopp vid en våglängd på 260 nm.Enligt Lambert-Beers lag kan vi beräkna RNA-koncentrationen från absorptionstoppen vid 260nm.Dessutom kan vi också beräkna renheten av RNA enligt förhållandet mellan 260nm, 280nm och 230nm absorptionstoppar.280nm och 230nm är absorptionstopparna för proteiner respektive små molekyler.Förhållandet mellan A260/A280 och A260/A230 av kvalificerad RNA-renhet bör vara större än 2. Om det är mindre än 2 betyder det att det finns protein- eller småmolekyler i RNA-provet och måste renas igen.Kontamineringskällor kommer att påverka nedströmsexperiment, såsom inhibering av amplifieringseffektiviteten av PCR-reaktioner, vilket resulterar i felaktiga kvantitativa resultat.Renheten hos RNA har stor inverkan på efterföljande resultat, så spektrofotometri är i allmänhet en oumbärlig kvalitetskontrolllänk i det första steget i nukleinsyraexperiment.
Figur 1. Typiskt RNA/DNA-absorptionsspektrum
02 Agarosgelelektrofores
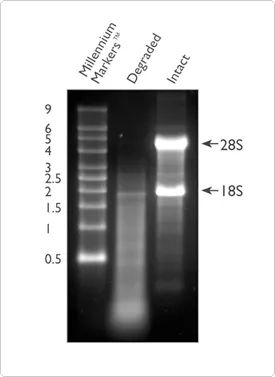
Förutom renhet är integriteten hos RNA också en av de viktiga indikatorerna för att bedöma kvaliteten på RNA.Nedbrytningen av RNA kommer att leda till ett stort antal korta fragment i provet, så antalet RNA-fragment som effektivt kan detekteras och täckas av referenssekvensen kommer att minska.RNA-integritet kan kontrolleras genom elektrofores av totalt RNA på en 1% agarosgel.Den här metoden kan konfigurera gelen själv eller använda det prefabricerade E-Gel™-systemet för integritetstestning.Mer än 80 % av det totala RNA är ribosomalt RNA, varav majoriteten består av 28S och 18S rRNA (i däggdjurssystem).RNA av god kvalitet kommer att visa två tydliga ljusa staplar, som är 28S respektive 18S ljusa staplar, vid 5 Kb och 2 Kb, och förhållandet tenderar att vara nära 2:1.Om det är i ett diffust tillstånd betyder det att RNA-provet kan ha brutits ned, och det rekommenderas att använda metoden som beskrivs senare för att ytterligare testa kvaliteten på RNA.
Figur 2. Jämförelse av nedbrutet (bana 2) och intakt RNA (bana 3) på agarosgelelektrofores
03 Agilent Bioanalyzer
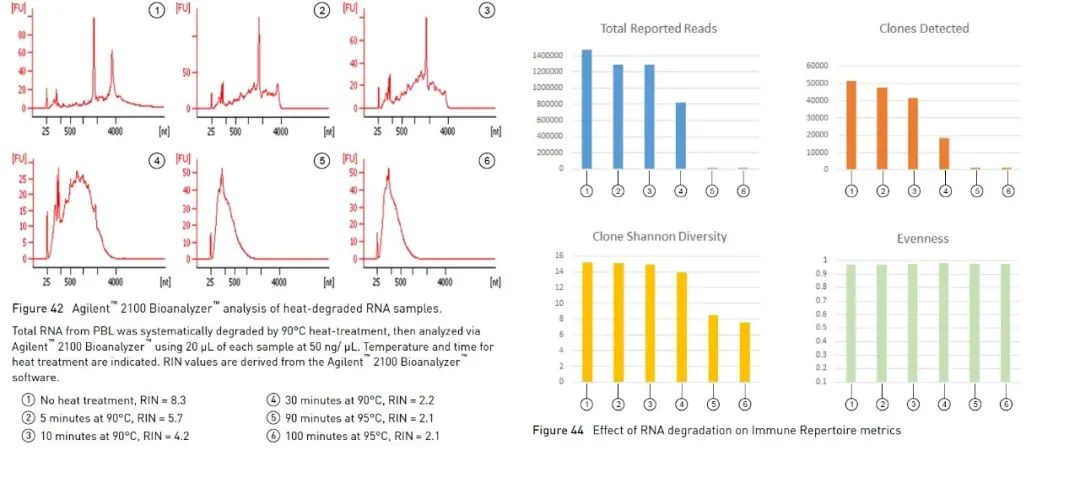
Förutom agarosgelelektroforesmetoden som beskrivs ovan, som kan hjälpa oss att identifiera integriteten av RNA enkelt och snabbt, kan vi också använda Agilent bioanalyzer för att bestämma RNA:s integritet.Den använder en kombination av mikrofluidik, kapillärelektrofores och fluorescens för att bedöma RNA-koncentration och integritet.Genom att använda den inbyggda algoritmen för att analysera profilen för RNA-provet kan Agilent-bioanalyzern beräkna ett referensvärde för RNA-integritet, RNA Integrity Number (nedan kallat RIN) [1].Ju större RIN-värdet är, desto högre är integriteten för RNA:t (1 är extremt försämrat, 10 är det mest kompletta).Vissa experiment som involverar RNA föreslår att man använder RIN som en parameter för kvalitetsbedömning.Om man tar sekvenseringsexperiment med hög genomströmning (hädanefter kallade NGS) som exempel, föreslår riktlinjerna för Oncomine™ Human Immune Repertoire, som används för att detektera B-cells- och T-cellsantigenreceptorer i Thermo Fishers Oncomine-panelserie, att prover med RIN-värden större än 4, effektivare avläsningar och kloningar kan mätas (Finesgure).Det finns olika rekommenderade intervall för olika paneler, och ofta kan ett högre RIN ge effektivare data.
Figur 3, i experiment med Oncomine™ Human Immune Repertoire, kan prover med RIN större än 4 detektera effektivare avläsningar och T-cellkloner.【2】
RIN-värdet har dock också vissa begränsningar.Även om RIN har en hög korrelation med kvaliteten på NGS experimentella data, är den inte lämplig för FFPE-prover.FFPE-prover har behandlats kemiskt under lång tid, och det extraherade RNA:t har i allmänhet ett relativt lågt RIN-värde.Detta betyder dock inte att experimentets effektiva data måste vara otillfredsställande.För att korrekt bedöma kvaliteten på FFPE-prover måste vi använda andra mätningar än RIN.Förutom RIN kan Agilent bioanalyzer även beräkna DV200-värdet som en utvärderingsparameter av RNA-kvalitet.DV200 är en parameter som beräknar andelen fragment större än 200 bp i ett RNA-prov.DV200 är en bättre indikator på FFPE-provkvalitet än RIN.För RNA som extraheras av FFPE har det en mycket hög korrelation med antalet gener som effektivt kan detekteras och mångfalden av gener [3].Även om DV200 kan kompensera för bristerna i kvalitetsdetekteringen av FFPE, kan Agilent bioanalyzer fortfarande inte heltäckande analysera kvalitetsproblemen i RNA-prover, inklusive om det finns inhibitorer i proverna.Inhibitorer i sig kan påverka amplifieringseffektiviteten i nedströmsexperiment och minska mängden användbar data.För att veta om det finns en hämmare i provet, kan vi anta den fluorescerande kvantitativa PCR-metoden i realtid som beskrivs härnäst.
04 fluorescerande kvantitativ PCR i realtid
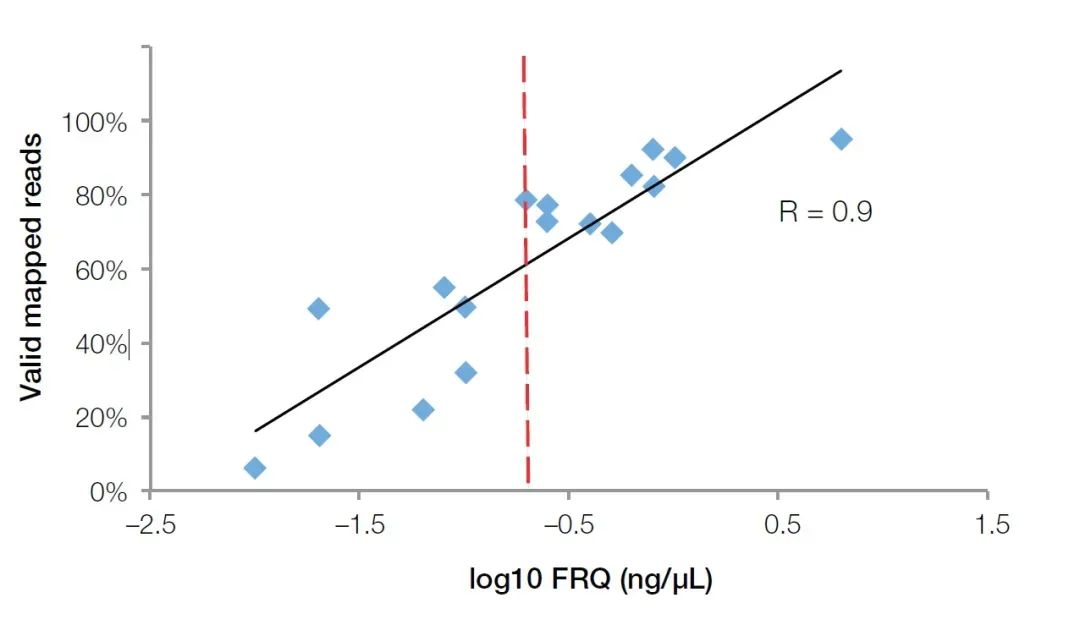
Den fluorescerande kvantitativa PCR-metoden i realtid kan inte bara detektera inhibitorerna i provet, utan också exakt återspegla kvaliteten på RNA i FFPE-provet.Jämfört med Agilent biologiska analysatorer är kvantitativa fluorescensinstrument i realtid mer populära i stora biologiska laboratorier på grund av deras bredare tillämpning.För att testa kvaliteten på RNA-prover behöver vi bara köpa eller förbereda primerprober för interna referensgener, såsom GUSB (Kat nr. Hs00939627).Genom att använda denna uppsättning primrar, prober och standarder (totalt RNA med känd koncentration) för att utföra absoluta kvantitativa experiment, kan den effektiva RNA-fragmentkoncentrationen beräknas som utvärderingsstandarden för RNA-kvalitet (Functional RNA Quantitation (FRQ) för kort).I ett NGS-test fann vi att FRQ för RNA-prover har en mycket hög korrelation med den effektiva datavolymen.För alla prover större än 0,2 ng/uL FRQ kan minst 70 % av avläsningarna effektivt täcka referenssekvensen (Figur 4).
Figur 4, FRQ-värdet som detekteras med den kvantitativa fluorescensmetoden har en mycket hög korrelation (R2>0,9) med de effektiva data som erhållits i NGS-experimentet.Den röda linjen är FRQ-värdet lika med 0,2 ng/uL (log10 = -0,7).【4】
Förutom att vara tillämplig på FFPE-prover kan den kvantitativa PCR-metoden i realtid också effektivt övervaka inhibitorer i prover.Vi kan lägga till provet som ska detekteras i reaktionssystemet med Internal Positive Control (IPC) och dess analys, och sedan utföra fluorescenskvantifiering för att erhålla Ct-värdet.Om Ct-värdet ligger efter Ct-värdet i reaktionen utan prov, indikerar det att inhibitorn finns i provet och hämmar amplifieringseffektiviteten i reaktionen.
05 Qubit fluorescerande färgningsmetod
Qubit Fluorometer är den mest använda lilla enheten för detektion av nukleinsyrakoncentration och renhet, som är lätt att använda och finns i nästan alla molekylärbiologiska laboratorier.Den beräknar noggrant koncentrationen av nukleinsyra genom att detektera och nukleinsyrabindande fluorescerande färgämne (Qubit detektionsreagens).Qubit har hög känslighet och specificitet och kan exakt kvantifiera RNA ned till pg/µL-koncentration.Förutom den välkända förmågan att noggrant kvantifiera nukleinsyrakoncentrationen, kan Thermo Fishers senaste nya modell, Qubit 4.0, även detektera integriteten hos RNA.Qubit 4.0:s RNA-detektionssystem (RNA IQ Assay) detekterar integriteten av RNA genom att samtidigt detektera två specifika fluorescerande färgämnen.Dessa två fluorescerande färgämnen kan binda till stora fragment respektive små fragment av RNA.Dessa två fluorescerande färgämnen indikerar andelen stora fragment av RNA i provet, och utifrån detta kan IQ-värdet (integritet och kvalitet) som representerar RNA-kvaliteten beräknas.IQ-värdet är tillämpligt på både FFPE- och icke-FFPE-prover och har stor inverkan på den efterföljande sekvenseringskvaliteten.Med NGS-experiment som exempel, i RNA-Seq-testexperimenten som utfördes på Ion torrent™-plattformen, hade de flesta prover med IQ-värden större än 4 minst 50 % effektiva avläsningar (Figur 5).Jämfört med de ovannämnda detektionsmetoderna är Qubit IQ Assay inte bara bekvämare att använda och tar mindre tid (inom fem minuter), utan har också en stor korrelation mellan det uppmätta parameterns IQ-värde och datakvaliteten i nedströmsexperiment.
Figur 5, det finns en stor korrelation mellan Qubit RNA IQ-värdet och de kartlagda läsningarna av RNA-Seq.【5】
Genom ovanstående introduktion tror jag att alla har en tillräcklig förståelse för olika RNA-kvalitetskontrollmetoder.I praktiken kan du välja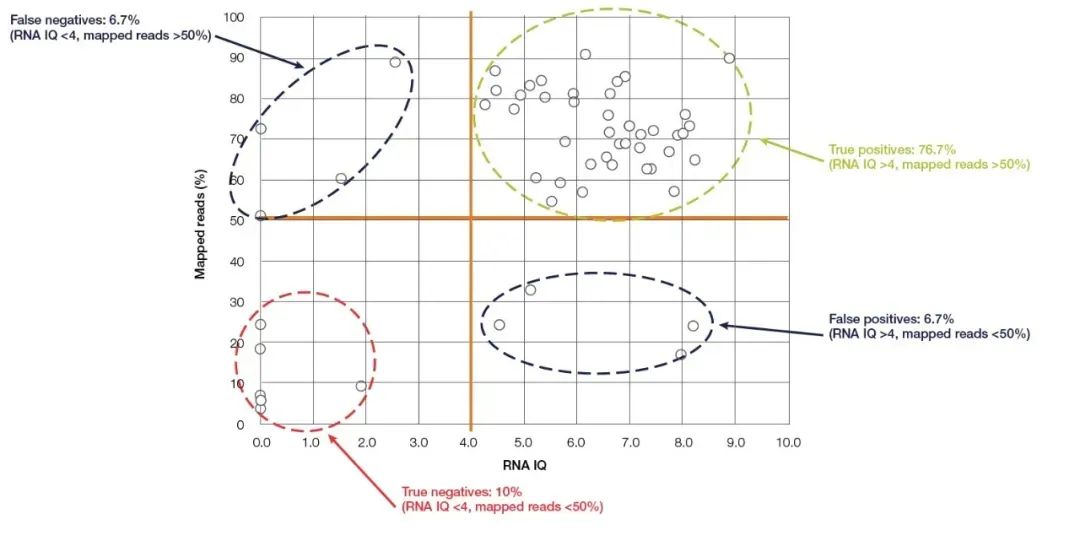 motsvarande metod enligt provtypen och befintliga instrument.Endast genom att kontrollera kvaliteten på RNA väl kan vi undvika misslyckanden i efterföljande experiment orsakade av dålig provkvalitet, vilket sparar dyrbar tid, energi och kostnader.
motsvarande metod enligt provtypen och befintliga instrument.Endast genom att kontrollera kvaliteten på RNA väl kan vi undvika misslyckanden i efterföljande experiment orsakade av dålig provkvalitet, vilket sparar dyrbar tid, energi och kostnader.
Referensprodukter:
Animal Total RNA Isolation Kit
referenser
【1】 Schroeder, A., Mueller, O., Stocker, S. et al.RIN: ett RNA-integritetsnummer för att tilldela integritetsvärden till RNA-mätningar.BMC Molecular Biol 7, 3 (2006).https:// doi .org/10.1186/1471-21 99-7-3
【2】 Användarhandbok för Oncomine Human Immune Repertoire (Pub. Nr. MAN0017438 Rev. C.0).
【3】Leah C Wehmas, Charles E Wood, Brian N Chorley, Carole L Yauk, Gail M Nelson, Susan D Hester, Enhanced Quality Metrics for Assessing RNA Derived From Archival Formalin-Fixed Paraffin-Embedded Tissue Samples, Toxicological Sciences, Volume 3–7ssue 170, August 7, Sue 170, Issue 170, Augustihttps://doi.org/10.1093/toxsci/
Posttid: 2023-jun-12