Utgångsmaterial: RNA
Kvantitativ omvänd transkriptions-PCR (RT-qPCR) är en experimentell metod som används i PCR-experiment med RNA som utgångsmaterial.I denna metod transkriberas först totalt RNA eller budbärar-RNA (mRNA) till komplementärt DNA (cDNA) genom omvänt transkriptas.Därefter utfördes en qPCR-reaktion med användning av cDNA som mall.RT-qPCR har använts i en mängd olika molekylärbiologiska tillämpningar, inklusive genuttrycksanalys, validering av RNA-interferens, mikroarrayvalidering, patogendetektion, genetisk testning och sjukdomsforskning.
Enstegs- och tvåstegsmetoder för RT-qPCR
RT-qPCR kan åstadkommas med en enstegs- eller tvåstegsmetod.Enstegs RT-qPCR kombinerar omvänd transkription och PCR-amplifiering, vilket gör att omvänt transkriptas och DNA-polymeras kan fullborda reaktionen i samma rör under samma buffertförhållanden.Enstegs RT-qPCR kräver endast användning av sekvensspecifika primrar.I tvåstegs RT-qPCR utförs omvänd transkription och PCR-amplifiering i två rör, med användning av olika optimerade buffertar, reaktionsförhållanden och primerdesignstrategier.
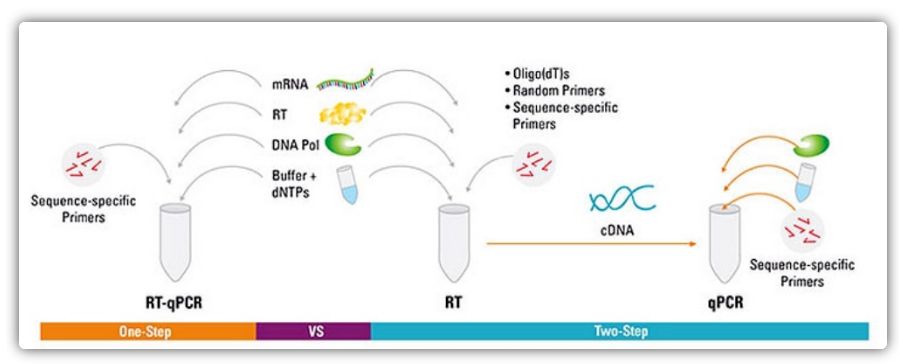
| Fördel | Nackdel | |
| Ett steg | Denna metod har mindre experimentella fel eftersom båda reaktionerna görs i ett rör
Färre pipetteringssteg minskar risken för kontaminering
Lämplig för förstärkning/screening med hög genomströmning, snabb och reproducerbar | Tvåstegsreaktioner kan inte optimeras separat
Eftersom reaktionsförhållandena äventyras genom att kombinera tvåstegsreaktionen är känsligheten inte lika bra som för tvåstegsmetoden
Antalet mål som detekteras av ett enda prov är litet |
| Två steg | Möjlighet att skapa stabila cDNA-bibliotek som kan lagras under långa tidsperioder och användas i flera reaktioner
Målgener och referensgener kan amplifieras från samma cDNA-bibliotek utan behov av flera cDNA-bibliotek
Reaktionsbuffertar och reaktionsförhållanden som möjliggör optimering av enstaka reaktionskörningar
Flexibelt val av triggervillkor | Användning av flera rör och fler pipetteringssteg ökar risken för DNA-kontamination, och tidskrävande.
Kräver mer optimering än enstegsmetoden |
Relaterade produkter:
RT-qPCR Easyᵀᴹ (One Step)-SYBR Grön I
RT-qPCR Easyᵀᴹ (One Step)-Taqman
RT Easyᵀᴹ I Master Premix för första-strängs CDNA-syntes
Real Time PCR Easyᵀᴹ-SYBR Green I Kit
Val av totalt RNA och mRNA
När du utformar ett RT-qPCR-experiment är det viktigt att bestämma om du ska använda totalt RNA eller renat mRNA som mall för omvänd transkription.Även om mRNA kan ge något högre känslighet, används totalt RNA fortfarande ofta.Anledningen till detta är att totalt RNA har en viktigare fördel som utgångsmaterial än mRNA.För det första kräver processen färre reningssteg, vilket säkerställer bättre kvantitativ återhämtning av mall och bättre normalisering av resultat till startcellsnummer.För det andra undviker den mRNA-anrikningssteget, vilket kan undvika möjligheten till sneda resultat på grund av olika återhämtning av olika mRNA.Sammantaget, eftersom i de flesta tillämpningar den relativa kvantifieringen av målgenen är viktigare än den absoluta känsligheten för detektionen, är totalt RNA mer lämpligt i de flesta fall.
Omvänd transkriptionsprimer
I tvåstegsmetoden kan tre olika metoder användas för att prima cDNA-reaktionen: oligo(dT)-primers, slumpmässiga primers eller sekvensspecifika primers.Typiskt används oligo(dT)-primrar och slumpmässiga primrar i kombination.Dessa primrar hybridiserar till mall-mRNA-strängen och ger omvänt transkriptas med en utgångspunkt för syntes.
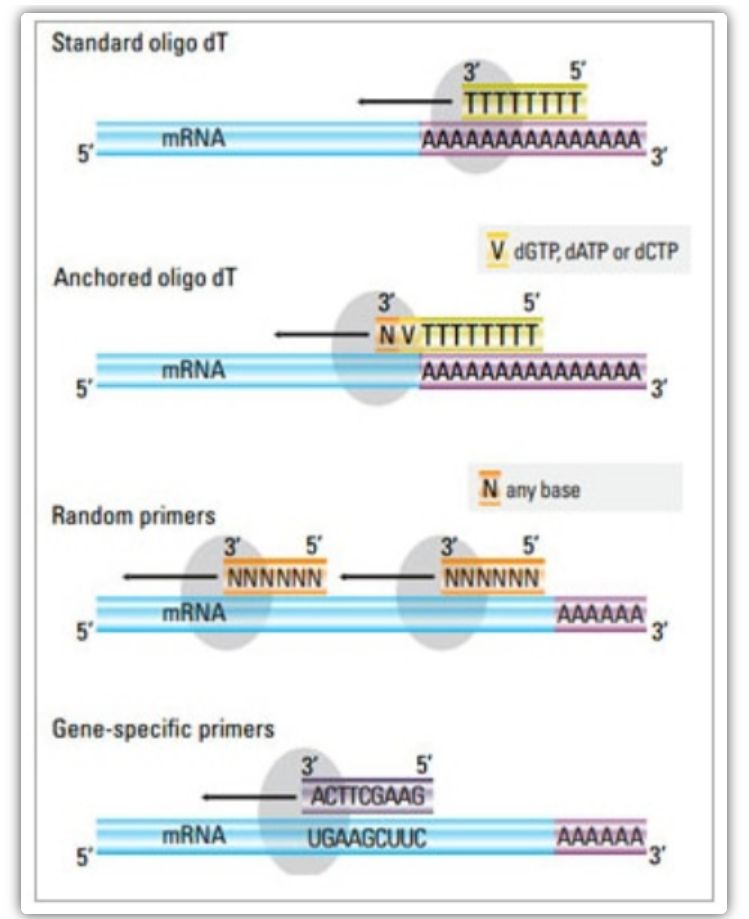
| Primerval | Struktur och funktion | Fördel | Nackdel |
| Oligo(dT)-primer (eller förankrad oligo(dT)-primer) | Förlängd hybridisering till tyminrester vid poly(A)-svansen av mRNA;ankaroligo(dT)-primer innehåller ett G, C eller A i 3′-änden (ankarställe) | Syntes av fullängds-cDNA från poly(A)-svansförsedd mRNA
Gäller när mindre utgångsmaterial finns tillgängligt
Förankringsstället säkerställer att oligo(dT)-primern binder till 5′ poly(A)-svansen av mRNA:t | Endast lämplig för amplifiering av gener med poly(A)-svansar
Erhåll cDNA trunkerat från primingstället*2 i poly(A)
Biased för att binda till 3′-änden*
*Denna möjlighet minimeras om förankrade oligo(dT)-primrar används |
| slumpmässig primer
| 6 till 9 baser i längd, vilket kan hybridisera till flera ställen under RNA-transkription | Anneal till alla RNA (tRNA, rRNA och mRNA)
Lämplig för transkript med betydande sekundär struktur, eller när mindre utgångsmaterial är tillgängligt
Högt cDNA-utbyte | cDNA transkriberas omvänt från allt RNA, vilket vanligtvis inte är önskvärt och kan späda ut signalen från mål-mRNA:t
få trunkerat cDNA |
| sekvensspecifika primrar | Anpassade primers riktade mot specifika mRNA-sekvenser | specifikt cDNA-bibliotek
Förbättra känsligheten
Använda omvända qPCR-primrar | Endast begränsad till syntesen av en enda målgen |
Omvänt transkriptas
Omvänt transkriptas är ett enzym som använder RNA för att syntetisera DNA.Vissa omvända transkriptaser har RNas-aktivitet och kan bryta ned RNA-strängar i RNA-DNA-hybridsträngar efter transkription.Om det inte har RNase-enzymatisk aktivitet kan RNaseH läggas till för högre qPCR-effektivitet.Vanligt använda enzymer inkluderar Moloney murint leukemivirus omvänt transkriptas och aviärt myeloblastomvirus omvänt transkriptas.För RT-qPCR är det idealiskt att välja ett omvänt transkriptas med högre termostabilitet, så att cDNA-syntes kan utföras vid högre temperaturer, vilket säkerställer framgångsrik transkription av RNA med högre sekundär struktur, samtidigt som deras fulla aktivitet bibehålls under hela reaktionen, vilket resulterar i högre cDNA-utbyten.
Relaterade produkter:
Foreasy M-MLV omvänt transkriptas
RNas H-aktivitet av omvänt transkriptas
RNaseH kan bryta ned RNA-strängar från RNA-DNA-duplexar, vilket möjliggör effektiv syntes av dubbelsträngat DNA.När lång mRNA används som mall kan RNA emellertid brytas ned i förtid, vilket resulterar i trunkerat cDNA.Därför är det ofta fördelaktigt att minimera RNaseH-aktivitet under cDNA-kloning om syntes av långa transkript önskas.Däremot är omvända transkriptaser med RNase H-aktivitet ofta fördelaktiga för qPCR-tillämpningar eftersom de förbättrar smältningen av RNA-DNA-duplex under den första PCR-cykeln.
Primer design
PCR-primrar som används för qPCR-steget i RT-qPCR bör helst utformas för att sträcka sig över en exon-exon-övergång, där en amplifieringsprimer potentiellt kan sträcka sig över en faktisk exon-intron-gräns.Eftersom intron-innehållande genomiska DNA-sekvenser inte amplifieras, minskar denna design risken för falska positiva amplifierade från kontaminerande genomiskt DNA.
Om primrar inte kan utformas för att separera exoner eller exon-exongränser, kan det vara nödvändigt att behandla RNA-prover med RNas-fritt DNas I eller dsDNas för att avlägsna genomisk DNA-kontamination.
RT-qPCR-kontroll
En negativ kontroll för omvänd transkription (-RT-kontroll) bör inkluderas i alla RT-qPCR-experiment för att detektera DNA-kontamination (som genomiskt DNA eller PCR-produkter från tidigare reaktioner).Denna kontroll innehåller alla reaktionskomponenter utom omvänt transkriptas.Eftersom omvänd transkription inte inträffar med denna kontroll, om PCR-amplifiering observeras, är kontaminering från DNA mest sannolikt.
Posttid: Aug-02-2022








