Detektions specificitet
I de flesta fall är syftet med primerdesign att maximera specificiteten för PCR.Detta bestäms av den mer eller mindre förutsägbara påverkan av många variabler.En viktig variabel är sekvensen vid 3′änden av primern.
Viktigt är att PCR-analyser utformade för specificitet är mer benägna att bibehålla hög effektivitet över ett brett dynamiskt område, eftersom analysen inte producerar ospecifika amplifieringsprodukter, och därmed konkurrerar med PCR-reagens eller hämmar den huvudsakliga amplifieringsreaktionen.
Naturligtvis är specificitet i vissa fall inte det viktigaste, till exempel när målet är att kvantifiera närbesläktade men det krävs olika patogener, speciell design, optimering och verifieringsstandarder.
Smältkurvan är en standardmetod för att bedöma specificiteten hos amplikoner, åtminstone när det gäller om ett enda mål ska amplifieras.Det måste dock betonas att smältkurvorna kan vara missvisande eftersom de till exempel kan påverkas av de kombinerade effekterna av suboptimala primers och låga mallkoncentrationer.
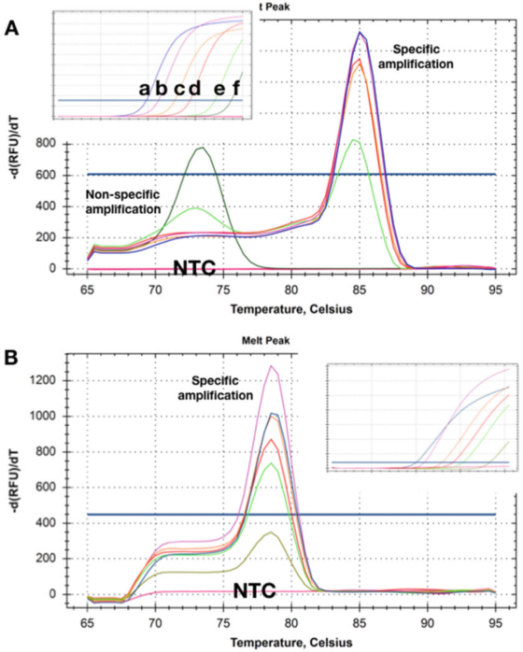
P5 |Smältkurvan visar Tm-skiften erhållna från två detektioner av olika mängder av två mål-DNA.
A. Vid högre koncentrationer (ad)) finns det ingen uppenbar primerdimer efter att qPCR-mätningen är klar.När mallkoncentrationen minskar till 50 exemplar (e), börjar en ospecifik produkt att dyka upp och blir den enda produkten med den lägsta koncentrationen (f).
B. Testet registrerade samma Tms vid alla målkoncentrationer, och det fanns ingen tydlig primerdimer även vid den lägsta koncentrationen (5 kopior).Vid användning av dessa två detektionsmetoder upptäcktes inga amplifieringsprodukter i NTC.
P5 visar upplösningskurvorna som erhållits med prover där mallen finns i olika koncentrationer.P 5a visar att vid de två lägsta koncentrationerna är Tms för de producerade icke-specifika amplifieringsprodukterna lägre än för de specifika amplikonerna.
Uppenbarligen kan denna detektionsmetod inte tillförlitligt användas för att detektera mål som finns i låga koncentrationer.
Intressant nog registrerade NTC, dvs prover utan DNA alls, inte (icke-specifika) amplifieringsprodukter, vilket indikerar att bakgrundsgenomiskt DNA kan delta i icke-specifik amplifiering/polymerisation.
Ibland kan sådana bakgrundsprimrar och ospecifik amplifiering inte åtgärdas, men det är ofta möjligt att designa en detektionsmetod som inte har ospecifik amplifiering i någon mallkoncentration och NTC (P 5b).
Här kommer även registrering av amplifieringen av målkoncentrationen med ett Cq på 35 att producera en specifik upplösningskurva.På liknande sätt visade NTC inga tecken på ospecifik amplifiering.Ibland kan detektionsbeteendet vara beroende av moderluten, och endast icke-specifik amplifiering detekteras i vissa buffertkompositioner, vilket kan vara relaterat till olika Mg2+-koncentrationer.
Detektionsstabilitet
Optimeringen av Ta är ett användbart steg i den empiriska verifierings- och optimeringsprocessen för qPCR-detektion.Den ger en direkt indikation på robustheten hos primeruppsättningen genom att visa den temperatur (eller temperaturintervallet) som ger den lägsta Cq utan att förstärka NTC.
Den två till fyra-faldiga skillnaden i känslighet kanske inte är viktig för personer med högt mRNA-uttryck, men för diagnostiska tester kan det betyda skillnaden mellan positiva och falskt negativa resultat.
Ta-egenskaperna hos qPCR-primrar kan variera mycket.Vissa tester är inte särskilt robusta, och om de inte utförs under det optimala Ta-värdet för primrarna kommer de snabbt att kollapsa.
Detta är viktigt eftersom denna typ av detektion ofta är problematisk i den verkliga världen, och provets renhet, koncentrationen av DNA eller närvaron av annat DNA kanske inte är optimal.
Dessutom kan antalet kopior variera inom ett brett intervall, och reagenserna, plastredskapen eller instrumenten kan skilja sig från de som används när testet ställs in.
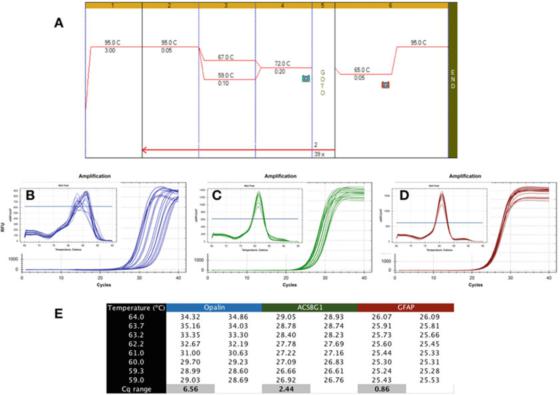
P6|Temperaturgradienten visar olika robusthet hos PCR-detektion.
A. Använd Biolines Sensifast SYBR mastermix (katalognummer BIO-98050) för att utföra PCR på cDNA framställt från RNA från mänsklig hjärna.
B. Använd Bio-Rads CFX qPCR-instrument för att registrera amplifieringskartan och upplösningskurvan för apalen (NM_033207, F: GCCATGGAGGAAAGTGACAGACC, R: CTCATGTGTGGGTGATCTCCTAGG).
C. Amplifieringsgraf och smältkurva för ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG).
D. Amplifieringsgraf och upplösningskurva för GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGATCTTC).
E. Cqs registrerade vid olika glödgningstemperaturer, som visar skillnaden i Cq registrerat under en 7C temperaturgradient.
P 6 visar ett typiskt resultat av ett oönskat test, där qPCR utfördes med en gradient Tas mellan 59C och 67C (P 6a), med användning av primers för tre mänskliga hjärnspecifika gener.
Det kan ses från amplifieringsgrafen att Opalin-primrar är långt ifrån idealiska eftersom deras optimala Ta-intervall är mycket smalt (Figur 6b), det vill säga Cqs är vitt spridda, vilket resulterar i att Cqs är signifikant jämfört med deras optimala Cqs Low.
Denna detektionsmetod är instabil och kan leda till suboptimal amplifiering.Därför bör detta primerpar göras om.Dessutom visar smältkurvanalysen (infälld) att specificiteten för denna detektionsmetod också kan vara problematisk, eftersom smältkurvan för varje Ta är olika.
ACSBG1-detektionsmetoden som visas i P 6c är mer robust än Opalin-detektionsmetoden ovan, men den är fortfarande långt ifrån idealisk, och det är troligt att den kan förbättras.
Vi betonar dock att det inte finns något nödvändigt samband mellan robusthet och specificitet, eftersom upplösningskurvan som produceras med denna detektionsmetod visar samma toppvärde i alla Tas (infälld).
Å andra sidan är robusthetstestet mycket mer tolerant, och producerar liknande Cqs i ett brett spektrum av Tas, som i GFAP-testet som visas i P 6d.
Skillnaden i Cqs som erhålls inom samma 8 grader Celsius-intervall är mindre än 1, och upplösningskurvan (infälld) bekräftar detektionsegenskaperna i detta temperaturområde.Det är värt att notera att det beräknade Tas och det faktiska Ta-intervallet kan vara mycket olika.
Det finns många riktlinjer utformade för att hjälpa forskare att designa effektiva primers, varav de flesta är baserade på sedan länge etablerade regler och mycket uppmärksamhet har ägnats åt 3′änden av primers.Det rekommenderas ofta att inkludera ett G eller C i 3'-änden och två G- eller C-baser (GC-klämma), men inte mer än två av de sista 5 baserna.
I praktiken kan dessa regler vägleda forskare, men de är inte nödvändigtvis korrekta under alla omständigheter.
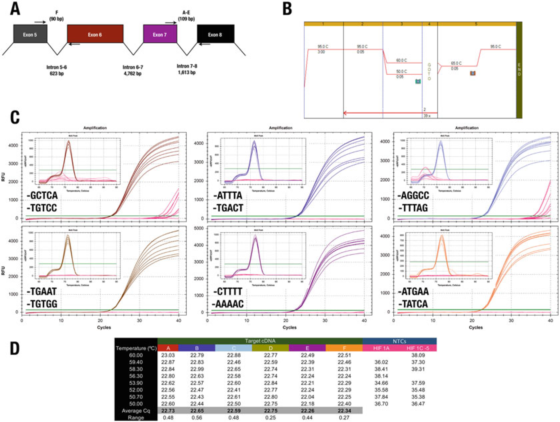
P7 |3'-änden av primern har liten effekt på specificitet eller effektivitet.
A. Positionen för primrarna för den humana HIF-la (NM_181054.2)-genen.
B. Använd Agilent Brilliant III SYBR Grön moderlut (kat. nr. 600882) för att förstärka sex testobjekt.
C. Amplifieringsgraf och smältkurva inspelad av Bio-Rads CFX qPCR-instrument och 3′end primers.NTC visas i rött.
D. Cqs post för varje testobjekt
Till exempel, resultatet i P 7 motsäger 3′end-regeln.Alla konstruktioner ger i princip samma resultat, med endast två primerkombinationer som leder till ospecifik amplifiering i NTC.
Vi kan dock inte stödja effekten av GC-klippet, för i det här fallet minskar inte specificiteten att använda A eller T som maximalt 30 baser.
Test C, där F-primern slutar i GGCC, registrerade Cqs i NTC, vilket indikerar att man kanske vill undvika dessa sekvenser i 30-änden.Vi betonar att det enda sättet att bestämma den bästa 3′-ändssekvensen för ett primerpar är att utvärdera några kandidatprimrar experimentellt.
Förstärkningseffektivitet
Viktigt, även om icke-specifik PCR-detektion aldrig kan bli specifik, kan amplifieringseffektiviteten justeras och maximeras på många olika sätt genom att ändra enzymet, moderluten, tillsatserna och cykliska förhållanden.
För att utvärdera effektiviteten av PCR-detektion är det bäst att använda en serieutspädning på 10 eller 5 gånger målnukleinsyran, det vill säga "standardkurvmetoden".
Om PCR-amplikoner eller syntetiska DNA-mål används för att generera en standardkurva, bör serieutspädningar av dessa mål blandas med en konstant mängd bakgrunds-DNA (som genomiskt DNA).
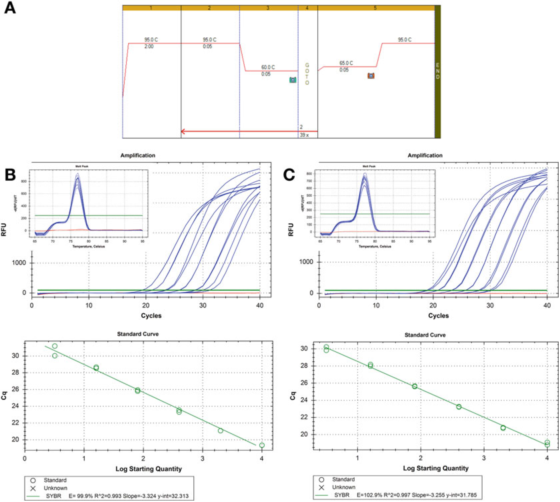
P8 |Spädningskurva för att utvärdera effektiviteten av PCR.
A. Använd primers för HIF-1: F: AAGAACTTTTAGGCCGCTCA och R: TGTCCTGTGGTGACTTGTCC och Agilents Brilliant III SYBR Green mastermix (katalognummer 600882) för PCR- och smältkurvförhållanden.
B. 100 ng RNA omvänt transkriberades, späddes 2 gånger och serieutspädda cDNA-prover späddes 5 gånger till 1 ng humant genomiskt DNA.Smältkurvan visas i infällningen.
C. RT-reaktionen, utspädningen och serieutspädningen upprepades för det andra cDNA-provet, och resultaten var liknande.
P 8 visar två standardkurvor, med användning av samma detektionsmetod på två olika cDNA-prover, resultatet är samma effektivitet, cirka 100 %, och R2-värdet är också liknande, det vill säga graden av passning mellan experimentdata och regressionslinjen eller data Grad av linjäritet.
De två standardkurvorna är jämförbara, men inte exakt lika.Om syftet är att korrekt kvantifiera målet, måste det noteras att det är oacceptabelt att tillhandahålla en kopianummerberäkning utan att förklara osäkerheten
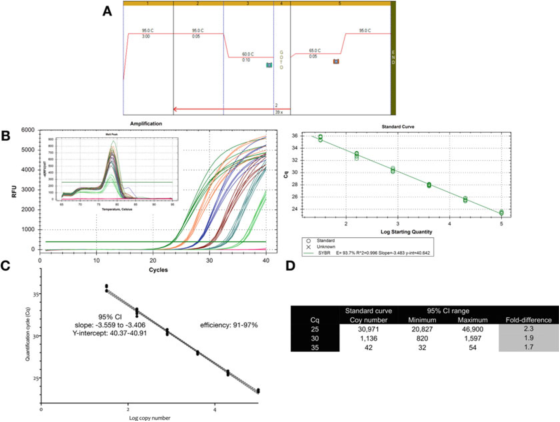
P9 |Mätosäkerhet förknippad med kvantifiering med hjälp av en standardkurva.
A. Använd primers för GAPDH (NM_002046) för att utföra PCR och smältkurvans förhållanden.F: ACAGTTGCCATGTAGACC och R: TAACTGGTTGAGCACAGG och Biolines Sensifast SYBR mastermix (katalognummer BIO-98050).
B. Amplifieringsdiagram, smältkurva och standardkurva registrerade med Bio-Rads CFX qPCR-instrument.
C. Standardkurvagraf och 95 % konfidensintervall (CI).
D. Kopiantalet och 95 % konfidensintervall för de tre Cq-värdena härledda från utspädningskurvan.
P 9 visar att för ett optimerat test är den inneboende variabiliteten för en enkel standardkurva ungefär 2 gånger (95 % konfidensintervall, minimum till maximum), vilket kan vara den minsta variabilitet som kan förväntas.
Relaterad produkt:
Posttid: 2021-09-30