Nyligen upptäckte jag något fantastiskt!Många avancerade experimentproffs runt honom kan inte ens några mycket grundläggande experimentella kunskapspunkter.
Kan du till exempel svara på följande frågor?
Är det någon skillnad mellan OD260 och A260?Vad betyder var och en?
OD är förkortningen för optisk densitet (optisk densitet), A är förkortningen av absorbans (absorbans), de två begreppen är egentligen desamma, "optisk densitet" är "absorbans", men "optisk densitet" är i linje med de flesta nationella standarder och mer standardiserade.
Vanligtvis mäter vi OD-värdet vid 260nm för att beräkna nukleinsyrakoncentrationen, så vad representerar 1OD?
Nukleinsyra har en maximal absorptionstopp vid en våglängd på 260nm, som innehåller både DNA och RNA, såväl som fragmenterade nukleinsyrafragment (detta är nyckelpunkten).
OD-värdet uppmätt vid en våglängd av 260 nm registrerades som OD260.Om provet är rent kan OD260-värdet beräkna koncentrationen av nukleinsyraprovet.
1 OD260=50 μg/ml dsDNA (dubbelsträngat DNA)
=37 μg/ml ssDNA (enkelsträngat DNA)
=40 μg/ml RNA
=30 μg/ml dNTPs (oligonukleotider)
Finns det någon koppling och skillnad mellan RT-PCR, Realtime-PCR och QPCR?
RT-PCR är en förkortning för Reverse Transcription PCR
Real Time PCR=qPCR, förkortning för Quantitative Real Time PCR
Även om realtids-PCR (realtidsfluorescerande kvantitativ PCR) och omvänd transkriptions-PCR (omvänd transkriptions-PCR) båda verkar vara förkortade som RT-PCR.Men den internationella konventionen är: RT-PCR hänvisar specifikt till omvänd transkriptions-PCR.
Vilka är de vanligaste nt, bp och kb för att beskriva längden av DNA/RNA i biologi?
nt = nukleotid
bp = baspar baspar
kb = kilobas
Naturligtvis skulle man säga att många inte bryr sig om dessa små detaljer!Alla gör det här, och ingen kommer att fråga dig vad det är.Du vet att det här är onödigt, eller hur?
Nej, nej, nej, det är mycket nödvändigt att veta detta!på grund av vilket?
För att du vill posta en artikel!Bror!Oavsett om du siktar på examen eller eftersträvar vetenskapliga forskningsresultat måste du lita på artiklar för att tala!
Nukleinsyraextraktion borde vara det enklaste och mest grundläggande experimentet.Kvaliteten på nukleinsyraextraktion avgör direkt resultaten av efterföljande experiment.
Även om jag har sagt det många gånger, finns det fortfarande många vänner som inte bryr sig.Den här gången bestämde jag mig för att flytta ur artikeln!

Minimiinformation för publicering av kvantitativa realtids-PCR-experiment, kallad MIQE, är en uppsättning riktlinjer för kvantitativa fluorescensexperiment som lanserats internationellt och som föreslår minimistandarder för den experimentella information som är nödvändig för att utvärdera kvantitativa PCR-experiment med fluorescens och för att publicera artiklar.Genom de experimentella förhållanden och analysmetoder som försöksledaren tillhandahåller kan granskarna bättre utvärdera giltigheten av forskarens experimentella schema.
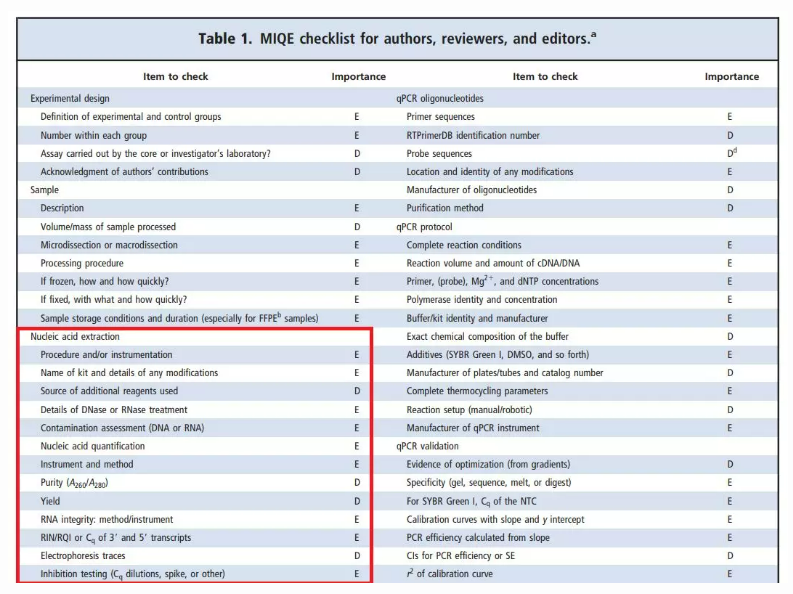
Det kan ses att i avsnittet om nukleinsyraextraktion har följande detektionsobjekt föreslagits,
"E" anger information som måste lämnas och "D" anger information som ska lämnas vid behov.
Formen är väldigt komplicerad, faktiskt vill jag säga att alla måste utgå från
renhet (D), utbyte (D), integritet (E) och konsistens (E) för att utvärdera nukleinsyror i dessa fyra aspekter.
Enligt experimentella vanor, prata först om utvärderingsmetoderna för renhet och koncentration.
OD-mätning är den favorit och enklaste detektionsmetoden för experimenterande.När det gäller principen kommer jag inte att gå in på detaljer här.Många laboratorier använder nu ultramikrospektrofotometrar för att direkt kvantitativt analysera nukleinsyraprover.Samtidigt som absorbansvärdet visas ger programmet direkt koncentrationsvärdet (nukleinsyra, protein och fluorescerande färgämne) och relaterade förhållanden.När det gäller analysen av OD-värdet, spara den här bilden så går det bra.
Universell OD-värdeslösningslista
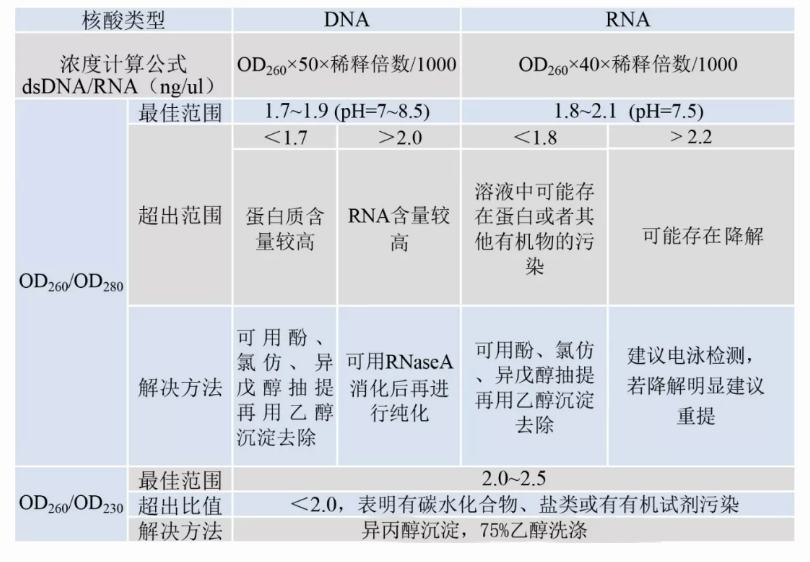 Det finns dock några varningar som måste tas fram separat för dig.
Det finns dock några varningar som måste tas fram separat för dig.
(Jag vet trots allt att ni måste vara de som sparar och väntar tills ni behöver dem!)
Not 1 Utrustning
OD-värdet kommer att påverkas av annan utrustning.Så länge som OD260 är inom ett visst område är värdena för OD230 och OD280 meningsfulla.Till exempel är absorbansintervallet för den vanliga Eppendorf D30 vid 260nm 0~3A, och NanoDrop One från Thermo är vid 260nm.Absorbansområdet 0,5~62,5A.
Anteckning 2Utspädningsreagens
OD-värdet kan påverkas av utspädning av olika reagens.Till exempel OD260/280-avläsningen av renat RNA i pH7,5 10 mM Trisbuffert är mellan 1,9-2,1, medan inneutral vattenlösningförhållandet kommer att vara lägre, kanske bara 1,8-2,0, men det betyder inte att kvaliteten på RNA förändras.
Anmärkning 3Återstående ämnen
Förekomsten av restsubstanser kommer att påverka noggrannheten i nukleinsyrakoncentrationsmätningen, så det är nödvändigt att undvika protein-, fenol-, polysackarid- och polyfenolrester i nukleinsyraprover så mycket som möjligt.
Men i själva verket är extraktion med organiska reagens en gammal metod.I kommersiella kit kan extraktionseffekten uppnås genom en silikabaserad adsorptionskolonn kombinerad med centrifugering, undvikande av giftiga och skadliga organiska reagens som är svåra att ta bort etc. Problemet, som t.ex.Foregenes nukleinsyraextraktionskit, använder inte DNas/RNas och giftiga organiska reagens under hela operationen, snabbt och säkert, och deneffekten ärBra(sade av misstag att den var flintskallig, men jag vet att du vill veta).
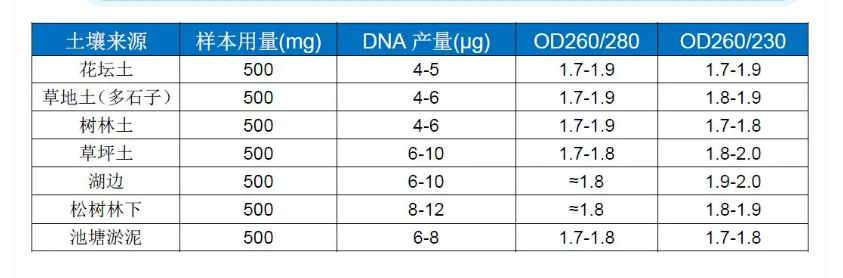
Exempel 1: Genomiskt DNA-extraktionsutbyte och renhet
Foregene Soil DNA Isolation Kit (DE-05511) behandlar jordprover från olika källor, och mängden och renheten av genomiskt DNA som erhålls visas i följande tabell:
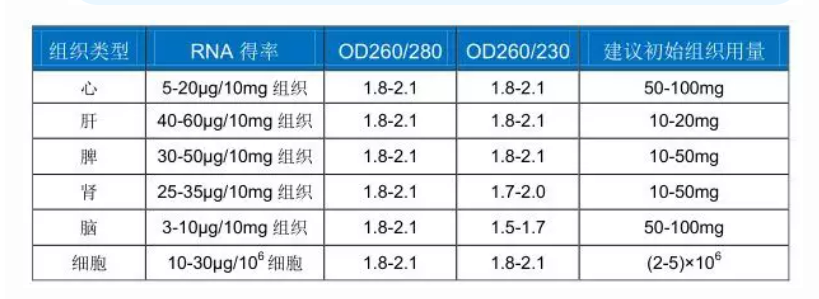
 Exempel 2: Vävnads-RNA-extraktionsutbyte och renhet
Exempel 2: Vävnads-RNA-extraktionsutbyte och renhet
Animal Total RNA Isolation Kit (RE-03012) bearbetade olika vävnadsprover, och mängden och renheten av RNA som erhållits visas i tabellen nedan (för musvävnad):
 Tro dock inte att du är klar med OD-värdet.Har du några ta hand om nyckelpunkterna jag ritade för dig i fronten?
Tro dock inte att du är klar med OD-värdet.Har du några ta hand om nyckelpunkterna jag ritade för dig i fronten?
Lägga märke till
Fragmenterade nukleinsyramolekyler kommer också att beräknas i absorbansen.Om du antar att du har genomiska DNA-rester i RNA:t kommer ditt OD-värde att tyckas vara mycket högt, men den faktiska koncentrationen av RNA kan inte fastställas.Om ditt RNA är Det är inte klart om det finns en nedbrytning, så vi behöver fortfarande en omfattande utvärderingsmetod för att ge en mer exakt bedömning, det vill säga utvärderingen av nukleinsyraintegritet som nämns i MIQE.
Posttid: 2022-jan-13








