Ⅰ. Öka reaktionssystemets känslighet:
1. Separera högkvalitativt RNA:
Framgångsrik cDNA-syntes kommer från högkvalitativt RNA.Högkvalitativt RNA bör säkerställa åtminstone en total längre och innehåller inte inhibitorer som inte innehåller registreringsenzymer, såsom EDTA eller SDS.Kvaliteten på RNA bestämmer det maximala värdet av sekvensinformationen som du kan transkribera till cDNA:t.Den allmänna RNA-reningsmetoden är en stegvis metod för användning av isoocyanat/acidofenol.För att förhindra förorening av RNas kräver RNA som separerats från ett prov rikt på RNas (som bukspottkörteln) lagring av formaldehyd för att spara högkvalitativt RNA, vilket är ännu mer så för långtidslagring.RNA extraherat från råttlevern bröts i princip ned efter en veckas förvaring i vatten, medan RNA extraherat från råttmjälten förblev stabil efter tre års förvaring i vatten.Dessutom är transkript större än 4kb mer känsliga för spår-RNas-nedbrytning än små transkript.För att öka stabiliteten hos lagrings-RNA-provet kan RNA:t lösas i en metalmamin av jon och lagras -70 °C.Tylid som används för att rädda RNA får inte innehålla ett annat föremål som bryter ned RNA.RNA, som härrör från bukspottkörteln, kan sparas i methalmamin i minst ett år.När du är redo att använda RNA kan du använda följande metoder för att fälla ut RNA: tillsätt NaCl till 0,2 m och 4 gånger volymen etanol, placera rumstemperatur i 3-5 minuter och 10 000 × g centrifugal i 5 minuter.
2. Använd omvänt transkriptas utan RNaseH-aktivitet (RNaseH-):
RNas-inhibitorer tillsätts ofta för omvända transkriptionsreaktioner för att öka längden och utbytet av cDNA-syntes.RNas-inhibitor tillsätts i den första kedjesyntesreaktionen i närvaro av buffertar och reduktionsmedel såsom DTT eftersom pre-cDNA-syntesprocessen denaturerar inhibitorn och frisätter därigenom bundna RNaser som bryter ned RNA.Protein RNas-hämmare förhindrar endast nedbrytningen av RNA av RNas A, B, C och förhindrar inte RNaser på huden, så man bör vara försiktig så att man inte introducerar RNaser från fingrarna trots användningen av dessa hämmare.
Omvänt transkriptas katalyserar omvandlingen av RNA till cDNA.Både M-MLV och AMV har endogen RNaseH-aktivitet förutom sin egen polymerasaktivitet.RNaseH-aktivitet konkurrerar med polymerasaktivitet för heterozygota strängar som bildas mellan RNA-mallar och DNA-primrar eller cDNA-förlängningssträngar, och bryter ned RNA: RNA-strängar i DNA-komplex.RNA-mallar som bryts ned av RNaseH-aktivitet kan inte längre användas som effektiva substrat för cDNA-syntes, vilket minskar utbytet och längden av cDNA-syntesen.Att eliminera eller kraftigt reducera RNaseH-aktivitet hos omvänt transkriptas skulle således vara till stor fördel.
SuperScriptⅡ omvänt transkriptas, MMLV omvänt transkriptas av RNaseH- och thermoScript omvänt transkriptas, AMV av RNaseH- gav mer fullängds cDNA än MMLV och AMV.RT-PCR-känslighet påverkas av mängden cDNA som syntetiseras.ThermoScript är mycket känsligare än AMV.Storleken på RT-PCR-produkter begränsas av förmågan hos omvänt transkriptas att syntetisera cDNA, speciellt vid kloning av större Cdna.Jämfört med MMLV ökade SuperScripⅡ signifikant utbytet av långa RT-PCR-produkter.RNaseH-s omvända transkriptas ökar också termisk stabilitet, så reaktionen kan utföras vid temperaturer högre än normalt på 37-42 ℃.Under de föreslagna syntesbetingelserna användes oligo(dT)-primrar och 10μCi [alfa-p]dCTP.Den totala produktionen av den första kedjan beräknades med TCA-fällningsmetoden.Fullängds-cDNA analyserades med användning av storlekssorterad remsa borttagning och räkning i en alkalisk agarosgel.
3. Öka värmekonserveringstemperaturen för omvänd transkription:
Högre hålltemperatur hjälper till att öppna den sekundära strukturen av RNA och öka utbytet av reaktionen.För de flesta RNA-mallar, att hålla RNA och primern vid 65°C utan buffert eller salt och sedan kyla dem snabbt på is eliminerar de flesta sekundära strukturer och tillåter primrarna att binda.Vissa mallar har dock fortfarande sekundär struktur, även efter termisk denaturering.Amplifiering av dessa svåra mallar kan utföras med användning av ThermoScript omvänt transkriptas och genom att placera omvänt transkriptasreaktionen vid högre temperaturer för att förbättra amplifieringen.Högre hålltemperaturer kan också öka specificiteten, särskilt när cDNA-syntes utförs med hjälp av genspecifika primrar (GSPS) (se kapitel 3).Om du använder GSP, se till att Tm-värdet för primern är detsamma som den förväntade hålltemperaturen.Använd inte oligo(dT) och slumpmässiga primrar över 60 ℃.Slumpmässiga primers måste hållas vid 25 ℃ i 10 minuter innan de ökar till 60 ℃.Förutom att använda högre temperaturer för omvänd transkription kan specificiteten förbättras genom att direkt överföra RNA/primerblandningen från 65℃ denatureringstemperaturen till den omvänd transkriptionshållningstemperaturen och tillsätta en förvärmd 2× reaktionsblandning (cDNA termisk initieringssyntes).Detta tillvägagångssätt hjälper till att förhindra den intermolekylära basparningen som sker vid lägre temperaturer.Att använda ett PCR-instrument förenklar de många temperaturomkopplarna som krävs för RT-PCR.
Det värmestabiliserade polymeraset fungerar som DNA-polymeras i närvaro av Mg2+ och RNA-polymeras i närvaro av Mn2+.Den kan hålla värme på upp till 65 ℃.Närvaron av Mn2+ under PCR minskar emellertid troheten, vilket gör Tth-polymeras mindre lämpligt för högprecisionsförstärkning, såsom cDNA-kloning.Dessutom är Tth mindre effektiv vid omvänd transkription, vilket minskar känsligheten, och eftersom ett enda enzym kan utföra omvänd transkription och PCR, kan kontrollreaktioner utan omvänd transkription inte användas för att skilja amplifierade produkter av cDNA från de av kontaminerat genomiskt DNA.
4. Tillsats som främjar omvänd transkription:
Tillsatsen av tillsatser, inklusive glycerin och DMSO, till den första kedjesyntesreaktionen kan minska stabiliteten hos nukleinsyrans dubbelsträng och avveckla den sekundära RNA-strukturen.Upp till 20 % glycerin eller 10 % DMSO kan tillsättas utan att påverka aktiviteten hos SuperScriptⅡ eller MMLV.AMV kan också tolerera upp till 20 % glycerol utan att minska aktiviteten.För att maximera känsligheten hos RT-PCR i SuperScriptⅡ omvänd transkriptionsreaktion kan 10 % glycerol tillsättas och isoleras vid 45 ℃.Om 1/10 av retrotranskriptionsreaktionsprodukten tillsätts till PCR, är koncentrationen av glycerol i amplifieringsreaktionen 0,4 %, vilket inte är tillräckligt för att hämma PCR.
5. RNaseH-bearbetning:
Känsligheten kan förbättras genom att behandla cDNA-syntesreaktioner med RNaseH före PCR.För vissa mallar tror man att RNA:t i cDNA-syntesreaktionen förhindrar bindningen av amplifierade produkter, i vilket fall RNaseH-behandlingen kan öka känsligheten.I allmänhet krävs RNaseH-behandling för amplifiering av en relativt lång cDNA-målmall i full längd, såsom tuberös scherosisⅡ med låg kopia.För denna svåra mall förbättrade RNaseH signalen som genererades av cDNA syntetiserat av SuperScriptⅡ eller AMV.För de flesta RT-PCR-reaktioner är RNaseH-behandlingen valfri eftersom det 95 ℃ isolerade PCR-denatureringssteget vanligtvis hydrolyserar RNA från RNA:DNA-komplexet.
6. Förbättrade metoder för att detektera små mängder RNA:
RT-PCR är särskilt utmanande när endast små mängder RNA är tillgängliga.Tillsatsen av glykogen som bärare under RNA-separation hjälper till att öka utbytet av små prover.Ett RNasfritt glykogen kan tillsättas samtidigt som Trizol.Glykogen är vattenlösligt och kan förbli i vattenfasen med RNA för att underlätta efterföljande utfällning.Den rekommenderade koncentrationen av RNas-fritt glykogen är 250 μg/ml för prover mindre än 50 mg vävnad eller 106 odlade celler.
Tillsatsen av acetylerad BSA för att omvända transkriptionsreaktioner med SuperScriptⅡ kan öka känsligheten, och för små mängder RNA kan en minskning av mängden SuperScriptⅡ och tillsats av 40 enheter RNaseOut nukleashämmare förbättra detektionsnivån.Om glykogen används vid RNA-separation, rekommenderas fortfarande tillägg av BSA- eller RNas-hämmare för att vända transkriptionsreaktioner med SuperScriptⅡ.
Ⅱ. Öka specificiteten för RT-PCR
1. cNDA-syntes:
Tre olika metoder kan användas för att initiera första strängens cDNA-syntes, och den relativa specificiteten för varje metod påverkar mängden och typen av syntetiserat cDNA.
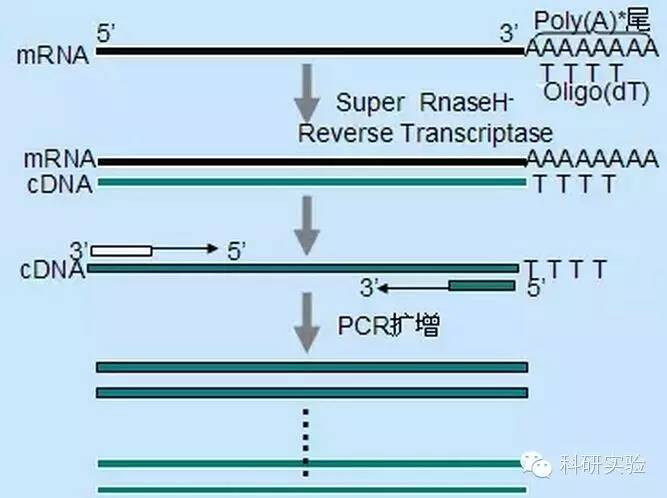
Den slumpmässiga primermetoden är den minst specifika av de tre metoderna.Primers hybridiseras vid flera ställen genom hela transkriptet för att producera kort cDNA med partiell längd.Denna metod används ofta för att erhålla 5′-terminala sekvenser och cDNA från RNA-mallar med sekundära strukturella regioner eller med terminerande ställen som omvänt transkriptas inte kan replikera.För att erhålla det längsta cDNA:t måste förhållandet mellan primers och RNA i varje RNA-prov bestämmas empiriskt.Den initiala koncentrationen av slumpmässiga primrar varierar från 50 till 250 ng per 20 μl reaktionssystem.Eftersom cDNA:t som syntetiseras från totalt RNA med användning av slumpmässiga primrar huvudsakligen är ribosomalt RNA, väljs i allmänhet poly(A)+RNA som mall.
Oligo(dT)-initiering är mer specifik än slumpmässiga primrar.Det hybridiserar med poly(A)-svansen som finns vid 3′-änden av mRNA i de flesta eukaryota celler.Eftersom poly(A)+RNA är ungefär 1 % till 2 % av totalt RNA, är mängden och komplexiteten av cDNA mycket mindre än om slumpmässiga primrar användes.På grund av dess höga specificitet kräver oligo(dT) i allmänhet inte optimering för RNA till primerförhållande och poly(A)+-selektion.Det rekommenderas att använda 0,5 μg oligo(dT) per 20 μl reaktionssystem.oligo(dT)12-18 är lämplig för de flesta RT-PCR.ThermoScript RT-PCR System ger oligo(dT)20 på grund av dess goda termiska stabilitet och är lämpligt för högre hålltemperaturer.
Genspecifika primrar (GSP) är de bästa specifika primrarna för omvänd transkriptionssteget.GSP är en antisens-oligonukleosid som specifikt kan hybridisera med RNA-destinationssekvenser, snarare än att hybridisera alla RNA som slumpmässiga primrar eller oligo(dT).Reglerna som används för att designa PCR-primers gäller även designen av omvänd transkriptionsreaktion GSP.GSP kan vara samma sekvens som amplifieringsprimern som hybridiseras i slutet av mRNA3', eller så kan GSP utformas för att hybridiseras nedströms med den omvända amplifieringsprimern.För vissa amplifierade objekt är det nödvändigt att designa mer än en antisens-primer för framgångsrik RT-PCR eftersom den sekundära strukturen av mål-RNA:t kan förhindra att primern binder.Det föreslås att använda 1 pmol antisense GSP i det första kedjesyntesreaktionssystemet på 20 μl.
2. Öka värmekonserveringstemperaturen för omvänd transkription:
För att dra full nytta av GSP-specificitet bör omvänt transkriptas med hög termisk stabilitet användas.Värmestabilt omvänt transkriptas kan isoleras vid högre temperaturer för att öka reaktionens stränghet.Till exempel, om en GSP hybridiseras vid 55°C, så utnyttjas inte specificiteten för GSP fullt ut om omvänd transkription utförs vid 37°C med låg rigoritet med användning av AMV eller M-MLV.Däremot kan SuperScripⅡ och ThermoScript reagera vid 50 ℃ eller högre, vilket eliminerar ospecifika produkter som produceras vid lägre temperaturer.För maximal specificitet kan RNA/primerblandningen överföras direkt från 65℃ denatureringstemperaturen till omvänd transkriptionshållningstemperatur med tillsats av en förvärmd 2 x reaktionsblandning (termisk initiering av cDNA-syntes).Detta hjälper till att förhindra basparning mellan molekyler vid låga temperaturer.Att använda ett PCR-instrument förenklar de många temperaturövergångar som krävs för RT-PCR.
3. Minska genomisk DNA-kontamination:
En potentiell svårighet med RT-PCR är att RNA kontaminerar genomiskt DNA.Användningen av bättre RNA-separationsmetoder, såsom Trizol Reagent, minskar genomisk DNA-kontamination i RNA-beredningar.För att undvika produkter som produceras från genomiskt DNA kan RNA:t behandlas med amplifieringsgrad DnasⅠ för att avlägsna kontaminerat DNA innan omvänd transkription.Proverna hölls vid 65 ℃ i 2,0 mM EDTA i 10 minuter för att avsluta DNaseⅠ-digestion.EDTA kelerar magnesiumjoner för att förhindra den magnesiumjonberoende RNA-hydrolysen som sker vid höga temperaturer.
För att separera amplifierat cDNA från genom-DNA-amplifieringsprodukten kan primrar som hybridiserar separat med det separerade exonet utformas.PCR-produkter härledda från cDNA kommer att vara kortare än de som härrör från kontaminerat genomiskt DNA.Ett kontrollerat experiment utan omvänd transkription utförs också på varje RNA-mall för att bestämma om ett givet fragment är från genomiskt DNA eller cDNA.PCR-produkter erhållna i frånvaro av omvänd transkription härleds från genomet.
Relaterad produkt
-Enstegssatsen gör att omvänd transkription och PCR kan utföras i samma rör.Den behöver bara lägga till mall-RNA, specifika PCR-primrar och RNase-fri ddH2O.
- Kvantitativ realtidsanalys av RNA kan utföras snabbt och exakt.
-Kitet använder ett unikt Foregene omvänd transkriptionsreagens och Foregene HotStar Taq DNA-polymeras kombinerat med ett unikt reaktionssystem för att effektivt förbättra amplifieringseffektiviteten och specificiteten hos reaktionen.
-Det optimerade reaktionssystemet gör att reaktionen har högre detektionskänslighet, starkare termisk stabilitet och bättre tolerans.
RT Easy II(Med GDNase) Master Premix för första-strängs CDNA-syntes för realtids-PCR med GDNase
-Effektiv förmåga att ta bort gDNA, vilket kan ta bort gDNA i mallen inom 2 minuter.
-Effektivt omvänt transkriptionssystem, det tar bara 15 minuter att slutföra syntesen av den första strängens cDNA.
-Komplexa mallar: mallar med högt GC-innehåll och komplex sekundär struktur kan också vändas med hög effektivitet.
-Högkänsligt omvänt transkriptionssystem, mallar på pg-nivå kan också få cDNA av hög kvalitet.
- Det omvända transkriptionssystemet har hög termisk stabilitet, den optimala reaktionstemperaturen är 42 ℃, och det har fortfarande bra omvänd transkriptionsprestanda vid 50 ℃.
Posttid: Mar-07-2023