- PCR är en metod som används för att amplifiera DNA från en liten mängd DNA-mall.RT-PCR använder omvänd transkription för att producera en DNA-mall från en RNA-källa som sedan kan amplifieras.
- PCR och RT-PCR är vanligtvis slutpunktsreaktioner, medan qPCR och RT-qPCR använder kinetiken för produktsynteshastigheten under PCR-reaktionen för att kvantifiera mängden närvarande mall.
- Nyare metoder, såsom digital PCR, ger absolut kvantifiering av den initiala DNA-mallen, medan metoder som isotermisk PCR minskar behovet av dyr utrustning för att ge tillförlitliga resultat.
Polymeraskedjereaktion (PCR) är en relativt enkel och allmänt använd molekylärbiologisk teknik för att amplifiera och detektera DNA- och RNA-sekvenser.Jämfört med traditionella metoder för DNA-kloning och amplifiering, som ofta kan ta dagar, kräver PCR bara några timmar.PCR är mycket känslig och kräver minimal mall för detektion och amplifiering av specifika sekvenser.Grundläggande PCR-metoder har avancerat ytterligare från enkel DNA- och RNA-detektion.Nedan har vi gett en översikt över de olika PCR-metoderna och de reagenser vi tillhandahåller på Enzo Life Sciences för dina forskningsbehov.Vi strävar efter att hjälpa forskare att snabbt få tillgång till PCR-reagenser att använda i deras nästa forskningsprojekt!
PCR
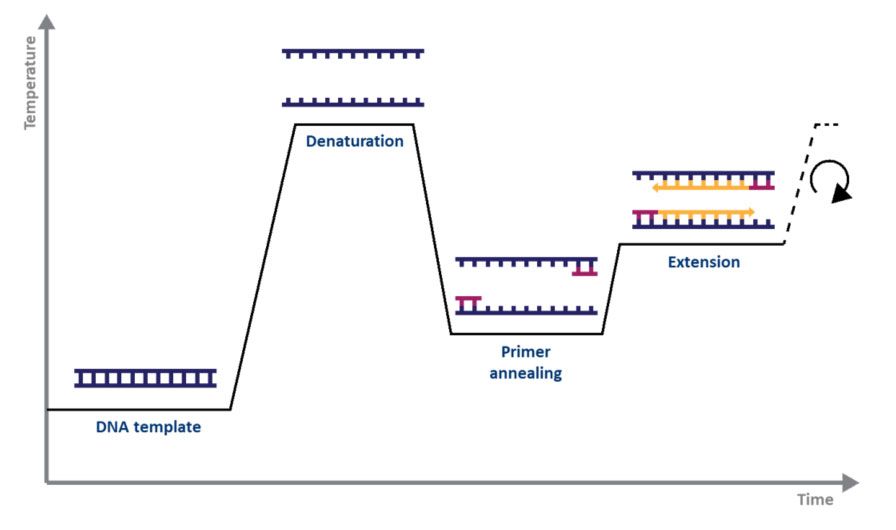
För standard PCR är allt du behöver ett DNA-polymeras, magnesium, nukleotider, primers, DNA-mallen som ska amplifieras och en termocykler.PCR-mekanismen är lika enkel som dess syfte: 1) dubbelsträngat DNA (dsDNA) är värmedenaturerat, 2) primrar anpassar sig till de enkla DNA-strängarna och 3) primrarna förlängs med DNA-polymeras, vilket resulterar i två kopior av ursprungliga DNA-strängen.Denaturerings-, hybridiserings- och förlängningsprocessen över en serie temperaturer och tider är känd som en amplifieringscykel (Fig. 1).
| Figur 1.Schematisk representation av en amplifieringscykel med PCR. |
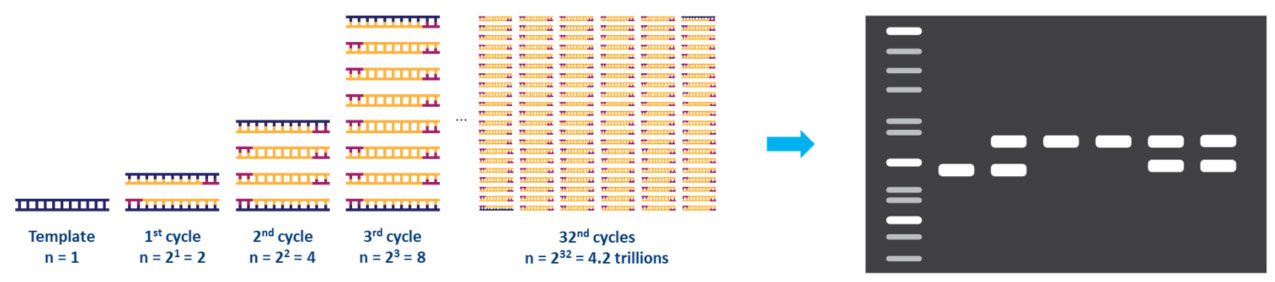
Varje steg i cykeln bör optimeras för mallen och primeruppsättningen som används.Denna cykel upprepas ungefär 20-40 gånger, och den amplifierade produkten kan sedan analyseras, typiskt med agarosgel (Fig. 2).
| Figur 2.Amplifiering av en DNA-mall med PCR och analys med agarosgelelektrofores. |
Eftersom PCR är en mycket känslig metod och mycket små volymer krävs för enstaka reaktioner, rekommenderas beredning av en mastermix för flera reaktioner.Mastermixen måste blandas väl och sedan delas efter antalet reaktioner, vilket säkerställer att varje reaktion kommer att innehålla samma mängd enzym, dNTPs och primers.Många leverantörer, som Enzo Life Sciences, erbjuder även PCR-blandningar som redan innehåller allt utom primers och DNA-mallen.
Guanin/Cytosin-rika (GC-rika) regioner representerar en utmaning i standard PCR-tekniker.GC-rika sekvenser är mer stabila än sekvenser med lägre GC-innehåll.Vidare tenderar GC-rika sekvenser att bilda sekundära strukturer, såsom hårnålsslingor.Som ett resultat är GC-rika dubbelsträngar svåra att fullständigt separera under denatureringsfasen.Följaktligen kan DNA-polymeras inte syntetisera den nya strängen utan hinder.En högre denatureringstemperatur kan förbättra detta, och justeringar mot en högre hybridiseringstemperatur och kortare hybridiseringstid kan förhindra den ospecifika bindningen av GC-rika primers.Ytterligare reagens kan förbättra amplifieringen av GC-rika sekvenser.DMSO, glycerol och betain hjälper till att störa de sekundära strukturerna som orsakas av GC-interaktioner och därigenom underlätta separationen av dubbelsträngarna.
Hot Start PCR
Ospecifik amplifiering är ett problem som kan uppstå under PCR.De flesta DNA-polymeraser som används för PCR fungerar bäst vid temperaturer runt 68°C till 72°C.Enzymet kan dock även vara aktivt vid lägre temperaturer, dock i lägre grad.Vid temperaturer långt under hybridiseringstemperaturen kan primers binda ospecifikt och leda till ospecifik amplifiering, även om reaktionen sätts upp på is.Detta kan förhindras genom att använda polymerashämmare som dissocierar från DNA-polymeraset först när en viss temperatur uppnåtts, därav termen varmstart-PCR.Inhibitorn kan vara en antikropp som binder polymeraset och denaturerar vid den initiala denatureringstemperaturen (typiskt 95°C).
High Fidelity Polymerase
Medan DNA-polymeraser amplifierar ganska exakt till den ursprungliga mallsekvensen, kan misstag i nukleotidmatchning inträffa.Felmatchningar i applikationer såsom kloning kan resultera i trunkerade transkript och felöversatta eller inaktiva proteiner nedströms.För att undvika dessa felmatchningar har polymeraser med en "korrekturläsande" aktivitet identifierats och införlivats i arbetsflödet.Det första korrekturläsande polymeraset, Pfu, identifierades 1991 i Pyrococcus furiosus.Detta Pfu-enzym har en 3' till 5' exonukleasaktivitet.När DNA:t amplifieras tar exonukleaset bort felmatchade nukleotider vid 3'-änden av strängen.Den korrekta nukleotiden ersätts sedan och DNA-syntesen fortsätter.Identifieringen av felaktiga nukleotidsekvenser baseras på bindningsaffiniteten för det korrekta nukleosidtrifosfatet med enzymet, där ineffektiv bindning saktar ner syntesen och möjliggör korrekt ersättning.Korrekturläsningsaktiviteten hos Pfu-polymeras resulterar i färre fel i den slutliga sekvensen jämfört med Taq DNA-polymeras.Under senare år har andra korrekturläsande enzymer identifierats och modifieringar av det ursprungliga Pfu-enzymet har gjorts för att ytterligare minska felfrekvensen under DNA-amplifiering.
RT-PCR
Omvänd transkriptions-PCR, eller RT-PCR, tillåter användning av RNA som mall.Ett ytterligare steg tillåter detektion och amplifiering av RNA.RNA:t omvänt transkriberas till komplementärt DNA (cDNA), med användning av omvänt transkriptas.Kvaliteten och renheten hos RNA-mallen är avgörande för framgången med RT-PCR.Det första steget av RT-PCR är syntesen av en DNA/RNA-hybrid.Omvänt transkriptas har också en RNas H-funktion, som bryter ned RNA-delen av hybriden.Den enkelsträngade DNA-molekylen kompletteras sedan av den DNA-beroende DNA-polymerasaktiviteten hos det omvända transkriptaset till cDNA.Effektiviteten av reaktionen med den första strängen kan påverka amplifieringsprocessen.Från och med nu används standard-PCR-förfarandet för att amplifiera cDNA:t.Möjligheten att omvandla RNA till cDNA genom RT-PCR har många fördelar, och den används främst för genuttrycksanalys.RNA är enkelsträngat och mycket instabilt, vilket gör det utmanande att arbeta med.Det fungerar vanligtvis som ett första steg i qPCR, som kvantifierar RNA-transkript i ett biologiskt prov.
qPCR och RT-qPCR
Kvantitativ PCR (qPCR) används för att detektera, karakterisera och kvantifiera nukleinsyror för många tillämpningar.I RT-qPCR kvantifieras RNA-transkript ofta genom att omvänd transkribera dem till cDNA först, som beskrivits ovan, och sedan utförs qPCR därefter.Som i standard PCR amplifieras DNA genom tre upprepade steg: denaturering, hybridisering och förlängning.Men i qPCR möjliggör fluorescerande märkning insamling av data när PCR fortskrider.Denna teknik har många fördelar på grund av det utbud av metoder och kemi som finns tillgängliga.
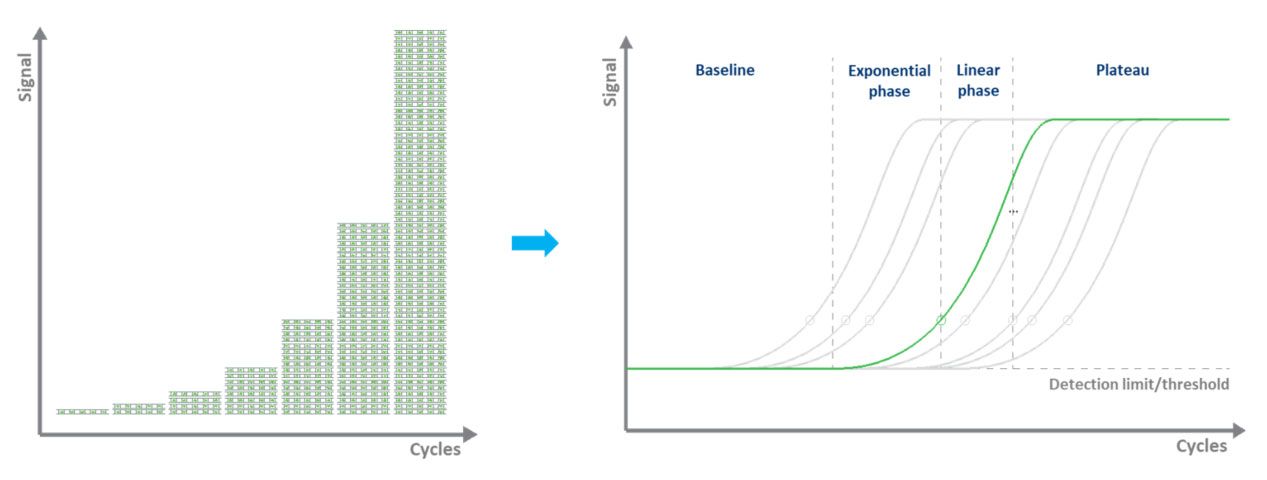
I färgämnesbaserad qPCR (vanligtvis grön) tillåter fluorescerande märkning kvantifiering av de amplifierade DNA-molekylerna genom att använda ett dsDNA-bindande färgämne.Under varje cykel mäts fluorescensen.Fluorescenssignalen ökar proportionellt mot mängden replikerat DNA.Därför kvantifieras DNA:t i "realtid" (Fig. 3).Nackdelarna med färgämnesbaserad qPCR är att endast ett mål kan undersökas åt gången och att färgämnet kommer att binda till eventuellt ds-DNA som finns i provet.
| Figur 3.Amplifiera en DNA-mall med qPCR och mäta fluorescenssignalen i realtid. |
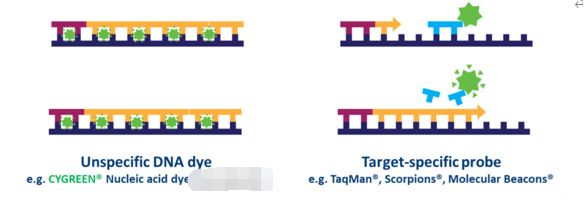
I sondbaserad qPCR kan många mål detekteras samtidigt i varje prov, men detta kräver optimering och design av en eller flera målspecifika prob(er) som används utöver primers.Flera typer av probdesigner finns tillgängliga, men den vanligaste typen är en hydrolysprob, som innehåller en fluorofor och quencher.Fluorescensresonansenergiöverföring (FRET) förhindrar emission av fluoroforen via quenchern medan sonden är intakt.Under PCR-reaktionen hydrolyseras emellertid sonden under primerförlängning och amplifiering av den specifika sekvens den är bunden till.Klyvningen av sonden separerar fluoroforen från quenchern och resulterar i en amplifieringsberoende ökning av fluorescens (fig. 4).Således är fluorescenssignalen från en probbaserad qPCR-reaktion proportionell mot mängden av probmålsekvensen som finns i provet.Eftersom sondbaserad qPCR är mer specifik än färgämnesbaserad qPCR, är det ofta den teknik som används i qPCR-baserade diagnostiska analyser.
| Figur 4.Skillnader mellan färgämnesbaserad och sondbaserad qPCR. |
Isotermisk förstärkning
PCR-teknikerna som nämns ovan kräver dyrbar termocyklisk utrustning för att exakt öka och sänka kammartemperaturerna för denaturerings-, glödgnings- och förlängningsstegen.Ett antal tekniker har utvecklats som inte behöver så exakta anordningar och som kan utföras i ett enkelt vattenbad eller till och med i cellerna av intresse.Dessa tekniker kallas gemensamt för isotermisk förstärkning och fungerar baserat på exponentiell, linjär eller kaskadförstärkning.
Den mest kända typen av isotermisk amplifiering är loop-medierad isoterm amplifiering, eller LAMP.LAMP använder exponentiell amplifiering vid 65⁰C för att amplifiera mall-DNA eller RNA.När man utför LAMP används fyra till sex primrar som är komplementära till regioner av mål-DNA:t med ett DNA-polymeras för att syntetisera nytt DNA.Två av dessa primrar har komplementära sekvenser som känner igen sekvenser i de andra primrarna och binder dem, vilket gör att en "loop"-struktur kan bildas i det nysyntetiserade DNA:t som sedan hjälper primer-annealing i efterföljande amplifieringsrundor.LAMP kan visualiseras med flera metoder, inklusive fluorescens, agarosgelelektrofores eller kolorimetri.Lättheten att visualisera och detektera närvaron eller frånvaron av produkt genom kolorimetri och bristen på dyr utrustning som krävs gjorde LAMP till ett lämpligt alternativ för SARS-CoV-2-testning i områden där kliniska laboratorietester inte var lättillgängliga, eller lagring och transport av prover inte var genomförbart, eller i labb som inte tidigare hade PCR-termocyklingutrustning.
Posttid: 2023-aug-19